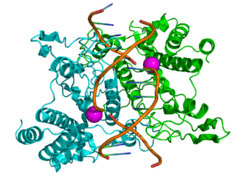
Restriksiyon enzimi
Restriksiyon enzimi veya restriksiyon endonükleazı çift zincirli DNA moleküllerindeki belli nükleotit dizilerini tanıyan ve her iki zinciri birlikte kesen bir enzim türüdür.[1][2][3] Bu özel enzimler, bakteri ve arkelerde bulunurlar ve virüslere karşı bir savunma mekanizmasına aittirler.[4][5] Konak bakteri hücresinde restriksiyon enzimleri seçici olarak yabancı DNA'ları keserler; konak DNA'yı restriksiyon enziminin etkinliğinden korunmak için bir değiştirme (modifikasyon) enzimi (bir metilaz) tarafından metillenir. Bu iki süreç toplu olarak restriksiyon modifikasyon sistemi olarak adlandırılır.[6] Bir restriksiyon enzimi DNA'yı kesmek için DNA çift sarmalının her şeker-fosfat omurgasından (yani her zincirden) birer kere olmak üzere iki kesme yapar.
Keşifleri
İlk restriksiyon enzimi HindIII'ün saflaştırılmasını[7] takiben pek çok başka restriksiyon enzimi keşfedilmiş ve karakterize edilmiştir.[8] 1978'de Daniel Nathans, Werner Arber ve Hamilton Smith restriksiyon enzimini keşiflerinden dolayı Nobel Tıp Ödülünü almışlardır.[9] Bu keşifleri rekombinant DNA teknolojisinin gelişimine öncülük etmiş, bunun sayesinde örneğin insülinin büyük miktarlarda üretimi için E. coli bakterisi kullanılabilmiştir.[10] 3000 üzerinde restriksiyon enzimi detaylı olarak çalışılmıştır, bunlardan 600'den fazlası ticari olarak elde edilebilir.[11] Bu enzimler laboratuvarlarda DNA modifikasyon ve maniplasyonlarında rutin olarak kullanılmaktadırlar.[12][13][14]
Tanıma bölgesi
5'-GTATAC-3' |
| Palindromik bir tanıma bölgesi ileri ve geri zincirlerde aynı biçimde okunur. |
Restriksiyon enzimleri spesifik bir nükleotit dizisi tanır [2] ve DNA'da çift zincirli bir kesik oluşturur. Tanıma dizilerinin uzunluğu 4 ila 8 nükleotit olup, çoğu palindromiktir, yani DNA'daki azotlu bazların dizisi ileri ve geri aynı okunur.[15] Teorik olarak DNA'da iki çeşit palindromik dizi olabilir. Yansımalı palindrom normal metinlerdeki gibi olur, aynı DNA dizisi üzerindeki dizinin normal ve tersten okunuşu aynı olur (örneğin GTAATG gibi). Evirtik (İng. inverted) tekrarlı palindrom da iki yönden aynı okunur ama ileri ve geri diziler komplemanter dizilerde yer alır. Örneğin GTATAC dizisinde olduğu gibi, bu dizinin komplemanter dizisi tersten okununca CATATG elde edilir.[16] Evirtik tekrarlar restriksiyon enzimlerinde daha yaygındır ve yansımalı palindromik dizilerden daha önemli biyolojik role sahiptir.
EcoRI retriksiyon enziminin yaptığı kesme "yapışkan" uçlar üretir,
![]()
buna karşın SmaI retriksiyon enziminin yaptığı kesme "küt" uçlar üretir
![]()
Her restriksiyon enzimi için DNA'daki tanıma bölgeleri farklıdır, kesim sonucu meydana gelen yapışkan ucun iplik uzantısının uzunluğu, dizisi ve zincir yönü (5' veya 3' yönünde) farklılıklar üretir.[17]
Aynı diziyi tanıyan farklı tanıma enzimleri neoşimerler olarak bilinir. Bunlar çoğunlukla diziyi iki farklı yerden keserler; eğer hem tanıma dizileri hem de kesme yerleri aynıysa bu enzimler izoşizomer olarak adalandırılır.
Bakteriler ürettikleri restriksiyon enzimlerinin kendi DNA'larını kesmemesi için, DNA metilazasyonu yoluyla nükleotitlerini değiştirerek (modifiye ederek) korurlar.[4]
Tipler
Restriksiyon endonükleazlar üç[18][19] veya dört[20][21][22] genel grupta kategorize edilirler (Tip I, II ve III), bileşenleri, enzim kofaktör gereksinimleri, hedef dizilerinin özellikleri ve DNA kesim yerinin hedef diziyle ilişkisine bağlı olarak.
Tip I
İlk keşfedilen restriksiyon enzimleri Tip I restriksiyon enzimleri olmuştur ve bunlar E. colinin iki farklı suşuna (K-12 ve B) özgüdürler.[23]
Bu enzimler tanıma bölgelerinden en azından 1000 baz çifti uzaklıktaki farklı bölgeleri keserler. Tanıma bölgesi asimetriktir ve 6-8 nükleotitlik bir boşlukla ayrılan iki kısımdan oluşur, biri 3-4 nükleotit içeren ve diğeri 4-5 nükleotit içeren. S-Adenozil metyonin (AdoMet), adenozin trifosfat (ATP) ve magnezyum iyonları (Mg2+) gibi birkaç enzim kofaktörü bu enzimlerin etkinliği için gereklidir.
Tip I restriksiyon enzimleri, HsdR, HsdM ve HsdS olarak adlandırılan üç altbirime sahiptirler; HsdR kesme için; HsdM konağın DNAsına metil grupları eklemek için; ve HsdS metiltransferaz etkinliğine ek olarak, tanıma bölgesinin kesim özgüllüğü için gereklidirler.[18][23]
Tip II
Tip II enzimler tip I enzimlerden birkaç yönden farklıdır. Tek tip proteinden oluşmuş dimer yapıya sahiptirler; tanıma bölgeleri genelde bölünmüş değildir, palindromiktir ve 4-8 nükleotit uzunluktadır; DNA'yı tanıdıkları ve kestikleri yer aynıdır; etkinlikleri için ATP veya AdoMet'e gerek göstermezler, kofaktör olarak genelde sadece Mg2+ gereksinimleri vardır. 1990'lar ve 2000'lerde bu enzim sınıfının tüm özelliklerin taşımayan yeni enzimler keşfedildiği için bu büyük enzim ailesini alt sınıflara ayıran yeni bir adlandırma sistemi geliştirildi.[15] Bu altgruplar bir sonek harf ile belirtilir.
Tip IIB restriksiyon enzimleri (örneğin BcgI and BplI) mültimeriktir, yani birden çok altbirimden oluşur.[15] DNA'yı tanıma dizisinin iki tarafından kesip çıkarırlar. Kofaktör olarak hem AdoMet hem de Mg2+ gereksinirler. Tip IIE restriksiyon endonükleazları (örneğin NaeI) tanıma dizilerinden iki kopyası ile etkileştikten sonra DNA'yı keserler.[15] Bir tanıma dizisi kesme hedefi olarak etkir, öbürü ie enzimin kesme verimini artıran, yani hızlandıran bir alosterik unsur olarak etkir. Tip IIF enzimler (örneğin NgoMIV) Tip IIE enzimlere benzer, onlar da tanıma dizilerinin iki kopyası ile etkileşir, ama ikisi birden keser.[15] Tip IIG enzimler (Eco57I gibi) tek bir altbirime sahiptir, klasik Tip II restriksiyon enzimleri gibi, ama etkin olmak için AdoMet kofaktörüne gerek duyarlar.[15] Tip IIM restriksiyon endonükleazları, DpnI gibi, metillenmiş DNA'yı tanıyıp kesebilirler.[15] Tip IIS restriksiyon enzimleri (FokI gibi) palindromik olmayan asimetrik tanıma dizilerinden beli bir uzaklıkta keserler.[15] Bu enzimler dimer olarak çalışabilir. Benzer olarak, Tip IIT restriksiyon enzimleri (örneğin Bpu10I ve BslI) iki farklı altbirimden oluşur. Bazıları palindromik dizileri tanır, bazılarının tanıma dizileri ise asimetriktir.[15]
Tip III
Tip III restriksiyon enzimleri (örneğin EcoP15) birbirine dönük olan iki ayrı, palindromik olmayan dizi tanırlar. DNA'yı tanıma yerinden 20-30 baz uzakta keserler.[25] Bu enzimler birden çok altbirime sahiptir; DNA metilasyonu ve restriksiyonu için, sırasıyla, AdoMet ve ATP kofaktörlerine gerek duyarlar.[26]
Tip IV
Tip IV restriksiyon enzimleri metillenmiş DNA'yı keser. Bunlar iki farklı altbirimden oluşur. DNA kesimi için Mg2 ve GTP kofaktör olarak gereklidir. Tanıma yeri iki parçalıdır. Metillenmiş bazlar arasında birden fazla kesim olur.[21]
Yapay Restriksiyon Enzimleri
Yapay restriksiyon enzimleri üretmek için doğal ve tasarımlı bir DNA bağlayıcı bölge ile bir nükleaz bölgesi (genelde FokI restriksiyon enziminin kesme bölgesi) birleştirilir.[27] Bu tür yapay restriksiyon enzimleri arzu edilen DNA dizilerini tanıyabilecek şekilde tasarlanabilir, ayrıca tanıma bölgelerinin uzunluğu 36 baz çifti uzunluğa varabilir.[28] Çinko parmak nükleazlar yapay restriksiyon enzimlerinin en yaygın kulanılanlarıdır, genelde genetik mühendislik[29][30][31][32] ve standart gen klonlama uygulamalarında da[33] Other artificial restriction enzymes are based on the DNA binding domain of TAL effectors.[34][35] kullanılırlar.
Adlandırma sistemi
| EcoRI adının türetilmesi | ||
|---|---|---|
| Kısaltma | Anlam | Açıklama |
| E | Escherichia | cins |
| co | coli | tür |
| R | RY13 | suş |
| I | İlk tespit edilmiş | O bakteride tespit edilme sırası |
1970'lerde keşfedilmelerinden beri çeşitli bakterilerde yüzlerce restriksiyon enzimi tespit edilmiştir. Her enzim elde edildiği bakteriye göre adlandırılır, bakterinin cinsi, türü ve suşuna dayalı bir adlandırma sistemine göre.[36][37] Örneğin EcoRI restriksiyon enziminin adı yandaki kutuda açıklandığı şekilde türetilmiştir.
Uygulamalar
Saflaştırılmış restriksiyon enzimleri çeşitli bilimsel uygulamalardaki DNA manipülasyonlarında kullanılır.
restriksiyon enzimleri gen klonlaması ve protein ifadesi deneylerinde, Plazmit vektörlerlerin içine genler sokmak için kullanilirlar. Gen klonlama deneylerinde kullanılan plazmitlerde genelde kısa bir "çoklu bağlayıcı" dizi (İng. polylinker; çoklu klonlama yeri) bulunur. Gen parçalarını plazmit vektörün içine sokarken bu diziler kolaylık sağlar; genin içinde doğal olarak bulunan restriksiyon yerleri DNA'yı kesmek için kullanılacak endonükleaz seçimini etkiler, çünkü arzu edilen DNA'ya zarar vermeden onun uçlarının kesilmesi gerekmektedir. Bir gen parçasının bir vektörün içine klonlamak için hem plazmit DNA'sı hem de gen parçası aynı restriksiyon enzimi ile kesilir, sonra bunlar DNA ligaz olarak adlandırılan bir enzimle birbirlerine yapıştırılır.[38][39]
Restriksiyon enzimleri DNA'da bulunan tek baz değişikliklerini (tek nükleotit polimorfizmleri, veya "SNP"leri) spesifik olarak tanıyarak gen alellerini ayırt etmekte kullanılırlar.[40][41] Bunun için o alelde bulunan bir restriksiyon yerinin bir SNP tarafından değişikliğe uğraması gerekmektedir. Bu yöntemle, bir DNA numunesini dizilemeden, bir retriksiyon enzimi ile onu genotiplemek mümkün olur. Numune önce DNA parçaları oluşturacak şekilde restrilksiyon enzimi ile sindirilir, sonra farklı büyüklükteki parçalar jel elektroforezi ile ayrıştırılır. Genelde, doğru restriksiyon yerine sahip olan aleller jelde iki görünür bant meydana getirir, değişlikliğe uğramış restriksiyon yeri olan parçalar ise kesilmezler ve sadece bir bant oluştururlar. Bant sayısı kişinin genotipini gösterir. Bu işlem bir restriksiyon haritalaması örneğidir.
Benzer şekilde, restriksiyon enzimleri Southern blot yöntemiyle genomik DNA'nın kesilmesinde kullanılır. Bu yöntem ile, bir kişinin genomunda bir genin kaç kopyası (veya paralogu) olduğu belirlenebilir. Bu yöntemin bir diğer uygulamasında belli bir toplulukta kaç tane gen mutasyonu (polimofizmi) olduğu belirlenebilir, buna restriksiyon parçası uzunluk polimorfizmi (İng. restriction fragment length polymorphism, RFLP) denir.[42]
Örnekler
- Daha çok ayrıntı için Restriksiyon enzimi kesme yerleri listesi maddesine bakınız.
Restriksiyon enzimi örnekleri:[43]
| Enzim | Kaynak | Tanıma yeri | Kesme |
|---|---|---|---|
| EcoRI | Escherichia coli |
5'GAATTC 3'CTTAAG |
5'---G AATTC---3' 3'---CTTAA G---5' |
| EcoRII | Escherichia coli |
5'CCWGG 3'GGWCC |
5'--- CCWGG---3' 3'---GGWCC ---5' |
| EcoRV* | Escherichia coli |
5'GATATC 3'CTATAG |
5'---GAT ATC---3' 3'---CTA TAG---5' |
| BamHI | Bacillus amyloliquefaciens |
5'GGATCC 3'CCTAGG |
5'---G GATCC---3' 3'---CCTAG G---5' |
| HindIII | Haemophilus influenzae |
5'AAGCTT 3'TTCGAA |
5'---A AGCTT---3' 3'---TTCGA A---5' |
| TaqI | Thermus aquaticus |
5'TCGA 3'AGCT |
5'---T CGA---3' 3'---AGC T---5' |
| NotI | Nocardia otitidis |
5'GCGGCCGC 3'CGCCGGCG |
5'---GC GGCCGC---3' 3'---CGCCGG CG---5' |
| HinfI | Haemophilus influenzae |
5'GANTCA 3'CTNAGT |
5'---G ANTC---3' 3'---CTNA G---5' |
| Sau3A | Staphylococcus aureus |
5'GATC 3'CTAG |
5'--- GATC---3' 3'---CTAG ---5' |
| PovII* | Proteus vulgaris |
5'CAGCTG 3'GTCGAC |
5'---CAG CTG---3' 3'---GTC GAC---5' |
| SmaI* | Serratia marcescens |
5'CCCGGG 3'GGGCCC |
5'---CCC GGG---3' 3'---GGG CCC---5' |
| HaeIII* | Haemophilus aegyptius |
5'GGCC 3'CCGG |
5'---GG CC---3' 3'---CC GG---5' |
| HgaI[44] | Haemophilus gallinarum |
5'GACGC 3'CTGCG |
5'---NN NN---3' 3'---NN NN---5' |
| AluI* | Arthrobacter luteus |
5'AGCT 3'TCGA |
5'---AG CT---3' 3'---TC GA---5' |
| EcoP15I | Escherichia coli |
5'CAGCAGN25NN 3'GTCGTCN25NN |
5'---CAGCAGN25NN ---3' 3'---GTCGTCN25 NN---5' |
| KpnI[45] | Klebsiella pneumoniae |
5'GGTACC 3'CCATGG |
5'---GGTAC C---3' 3'---C CATGG---5' |
| PstI[45] | Providencia stuartii |
5'CTGCAG 3'GACGTC |
5'---CTGCA G---3' 3'---G ACGTC---5' |
| SacI[45] | Streptomyces achromogenes |
5'GAGCTC 3'CTCGAG |
5'---GAGCT C---3' 3'---C TCGAG---5' |
| SalI[45] | Streptomyces albus |
5'GTCGAC 3'CAGCTG |
5'---G TCGAC---3' 3'---CAGCT G---5' |
| ScaI[45] | Streptomyces caespitosus |
5'AGTACT 3'TCATGA |
5'---AGT ACT---3' 3'---TCA TGA---5' |
| SpeI | Sphaerotilus natans |
5'ACTAGT 3'TGATCA |
5'---A CTAGT---3' 3'---TGATC A---5' |
| SphI[45] | Streptomyces phaeochromogenes |
5'GCATGC 3'CGTACG |
5'---GCATG C---3' 3'---C GTACG---5' |
| StuI[46][47] | Streptomyces tubercidicus |
5'AGGCCT 3'TCCGGA |
5'---AGG CCT---3' 3'---TCC GGA---5' |
| XbaI[45] | Xanthomonas badrii |
5'TCTAGA 3'AGATCT |
5'---T CTAGA---3' 3'---AGATC T---5' |
Anahtar:
* = küt uçlar
N = C, G, T veya A
W = A veya T
Ayrıca bakınız
- Bazı restriksiyon enzimleri hakkında ayrıntılı maddeler: EcoRV, HindIII, BglII.
- Restriksiyon enzimi kesme yerleri listesi
- Hedefli endonükleazları (homing endonükleazlar)
- Hedefli endonükleaz kesme yerleri listesi
- İzoşizomer.
- Yıldız etkinliği
- Moleküler ağırlık büyüklük belirteci
Kaynakça
- ↑ Roberts RJ (November 1976). "Restriction endonucleases". CRC Crit. Rev. Biochem. 4 (2): 123–64. PMID 795607.
- 1 2 Kessler C, Manta V (August 1990). "Specificity of restriction endonucleases and DNA modification methyltransferases a review (Edition 3)". Gene 92 (1-2): 1–248. DOI:10.1016/0378-1119(90)90486-B. PMID 2172084.
- ↑ Pingoud A, Alves J, Geiger R (1993). "Chapter 8: Restriction Enzymes". Burrell, Michael. Enzymes of Molecular Biology. 16. Totowa, NJ: Humana Press. s. pages 107-200. ISBN 0-89603-234-5.
- 1 2 Arber W, Linn S (1969). "DNA modification and restriction". Annu. Rev. Biochem. 38: 467–500. DOI:10.1146/annurev.bi.38.070169.002343. PMID 4897066.
- ↑ Krüger DH, Bickle TA (September 1983). "Bacteriophage survival: multiple mechanisms for avoiding the deoxyribonucleic acid restriction systems of their hosts". Microbiol. Rev. 47 (3): 345–60. PMC 281580. PMID 6314109. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=281580.
- ↑ Kobayashi I (September 2001). "Behavior of restriction-modification systems as selfish mobile elements and their impact on genome evolution". Nucleic Acids Res. 29 (18): 3742–56. DOI:10.1093/nar/29.18.3742. PMC 55917. PMID 11557807. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=55917.
- ↑ Roberts RJ (April 2005). "How restriction enzymes became the workhorses of molecular biology". Proc. Natl. Acad. Sci. U.S.A. 102 (17): 5905–8. DOI:10.1073/pnas.0500923102. PMC 1087929. PMID 15840723. http://www.pnas.org/cgi/pmidlookup?view=long&pmid=15840723.
- ↑ Danna K, Nathans D (December 1971). "Specific cleavage of simian virus 40 DNA by restriction endonuclease of Hemophilus influenzae". Proc. Natl. Acad. Sci. U.S.A. 68 (12): 2913–7. DOI:10.1073/pnas.68.12.2913. PMC 389558. PMID 4332003. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pubmed&pubmedid=4332003.
- ↑ "The Nobel Prize in Physiology or Medicine". The Nobel Foundation. 1978. 6 Nisan 2016 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20160406234641/http://www.nobelprize.org/nobel_prizes/medicine/laureates/1978/. Erişim tarihi: 2008-06-07.
- ↑ Villa-Komaroff L, Efstratiadis A, Broome S, Lomedico P, Tizard R, Naber SP, Chick WL, Gilbert W. (August 1978). "A bacterial clone synthesizing proinsulin". Proc. Natl. Acad. Sci. U.S.A. 75 (8): 3727–31. PMC 392859. PMID 358198. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=392859.
- ↑ Roberts RJ, Vincze T, Posfai J, Macelis D. (2007). "REBASE--enzymes and genes for DNA restriction and modification". Nucleic Acids Res 35 (Database issue): D269-70. DOI:10.1093/nar/gkl891. PMID 17202163. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pubmed&pubmedid=17202163.
- ↑ Primrose, Sandy B.; Old, R. W. (1994). Principles of gene manipulation: an introduction to genetic engineering. Oxford: Blackwell Scientific. ISBN 0-632-03712-1.
- ↑ Micklos, David A.; Bloom, Mark V.; Freyer, Greg A. (1996). Laboratory DNA science: an introduction to recombinant DNA techniques and methods of genome analysis. Menlo Park, Calif: Benjamin/Cummings Pub. Co. ISBN 0-8053-3040-2.
- ↑ Adrianne Massey; Helen Kreuzer (2001). Recombinant DNA and Biotechnology: A Guide for Students. Washington, D.C: ASM Press. ISBN 1-55581-176-0.
- 1 2 3 4 5 6 7 8 9 Pingoud A, Jeltsch A (September 2001). "Structure and function of type II restriction endonucleases". Nucleic Acids Res. 29 (18): 3705–27. DOI:10.1093/nar/29.18.3705. PMC 55916. PMID 11557805. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=55916.
- ↑ Molecular Biology: Understanding the Genetic Revolution, by David P. Clark. Elsevier Academic Press, 2005. ISBN 0-12-175551-7.
- ↑ Goodsell DS (2002). "The molecular perspective: restriction endonucleases". Stem Cells 20 (2): 190–1. PMID 11897876. http://stemcells.alphamedpress.org/cgi/pmidlookup?view=long&pmid=11897876.
- 1 2 Bickle TA, Krüger DH (June 1993). "Biology of DNA restriction". Microbiol. Rev. 57 (2): 434–50. PMC 372918. PMID 8336674. http://mmbr.asm.org/cgi/pmidlookup?view=long&pmid=8336674.
- ↑ Boyer HW (1971). "DNA restriction and modification mechanisms in bacteria". Annu. Rev. Microbiol. 25: 153–76. DOI:10.1146/annurev.mi.25.100171.001101. PMID 4949033.
- ↑ Yuan R (1981). "Structure and mechanism of multifunctional restriction endonucleases". Annu. Rev. Biochem. 50: 285–319. DOI:10.1146/annurev.bi.50.070181.001441. PMID 6267988.
- 1 2 Rao DN, Sistla S (2004). "S-Adenosyl-L-methionine-dependent restriction enzymes". Crit. Rev. Biochem. Mol. Biol. 39 (1): -. DOI:10.1080/10409230490440532. PMID 15121719.
- ↑ Williams RJ (2003). "Restriction endonucleases: classification, properties, and applications". Mol. Biotechnol. 23 (3): -. PMID 12665693.
- 1 2 Murray NE (June 2000). "Type I restriction systems: sophisticated molecular machines (a legacy of Bertani and Weigle)". Microbiol. Mol. Biol. Rev. 64 (2): 412–34. PMC 98998. PMID 10839821. http://mmbr.asm.org/cgi/pmidlookup?view=long&pmid=10839821.
- ↑ PDB 1qps Gigorescu A, Morvath M, Wilkosz PA, Chandrasekhar K, Rosenberg JM (2004). "The integration of recognition and cleavage: X-ray structures of pre-transition state complex, post-reactive complex, and the DNA-free endonuclease". Alfred M. Pingoud. Restriction Endonucleases (Nucleic Acids and Molecular Biology, Volume 14). Berlin: Springer. s. 137–178. ISBN 3-540-20502-0.
- ↑ Dryden DT, Murray NE, Rao DN (September 2001). "Nucleoside triphosphate-dependent restriction enzymes". Nucleic Acids Res. 29 (18): 3728–41. DOI:10.1093/nar/29.18.3728. PMC 55918. PMID 11557806. http://nar.oxfordjournals.org/cgi/pmidlookup?view=long&pmid=11557806.
- ↑ Meisel A, Bickle TA, Krüger DH, Schroeder C (January 1992). "Type III restriction enzymes need two inversely oriented recognition sites for DNA cleavage". Nature 355 (6359): 467–9. DOI:10.1038/355467a0. PMID 1734285.
- ↑ Kim YG, Cha J, Chandrasegaran S (February 1996). "Hybrid restriction enzymes: zinc finger fusions to Fok I cleavage domain". Proc. Natl. Acad. Sci. U.S.A. 93 (3): 1156–60. DOI:10.1073/pnas.93.3.1156. PMC 40048. PMID 8577732. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=40048.
- ↑ Urnov FD, Rebar EJ, Holmes MC, Zhang HS, Gregory PD (September 2010). "Genome editing with engineered zinc finger nucleases". Nat. Rev. Genet. 11 (9): 636–46. DOI:10.1038/nrg2842. PMID 20717154.
- ↑ Townsend JA, Wright DA, Winfrey RJ, Fu F, Maeder ML, Joung JK, Voytas DF (May 2009). "High-frequency modification of plant genes using engineered zinc-finger nucleases". Nature 459 (7245): 442–5. DOI:10.1038/nature07845. PMC 2743854. PMID 19404258. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=2743854.
- ↑ Shukla VK, Doyon Y, Miller JC, DeKelver RC, Moehle EA, Worden SE, Mitchell JC, Arnold NL, Gopalan S, Meng X, Choi VM, Rock JM, Wu YY, Katibah GE, Zhifang G, McCaskill D, Simpson MA, Blakeslee B, Greenwalt SA, Butler HJ, Hinkley SJ, Zhang L, Rebar EJ, Gregory PD, Urnov FD (May 2009). "Precise genome modification in the crop species Zea mays using zinc-finger nucleases". Nature 459 (7245): 437–41. DOI:10.1038/nature07992. PMID 19404259.
- ↑ Ekker SC (2008). "Zinc finger-based knockout punches for zebrafish genes". Zebrafish 5 (2): 121–3. DOI:10.1089/zeb.2008.9988. PMC 2849655. PMID 18554175. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=2849655.
- ↑ Geurts AM, Cost GJ, Freyvert Y, Zeitler B, Miller JC, Choi VM, Jenkins SS, Wood A, Cui X, Meng X, Vincent A, Lam S, Michalkiewicz M, Schilling R, Foeckler J, Kalloway S, Weiler H, Ménoret S, Anegon I, Davis GD, Zhang L, Rebar EJ, Gregory PD, Urnov FD, Jacob HJ, Buelow R (July 2009). "Knockout rats via embryo microinjection of zinc-finger nucleases". Science 325 (5939): 433. DOI:10.1126/science.1172447. PMC 2831805. PMID 19628861. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=2831805.
- ↑ Tovkach A, Zeevi V, Tzfira T (October 2010). "Expression, purification and characterization of cloning-grade zinc finger nuclease". J Biotechnol. DOI:10.1016/j.jbiotec.2010.10.071. PMID 21029755.
- ↑ Christian M, Cermak T, Doyle EL, Schmidt C, Zhang F, Hummel A, Bogdanove AJ, Voytas DF (October 2010). "Targeting DNA double-strand breaks with TAL effector nucleases". Genetics 186 (2): 757–61. DOI:10.1534/genetics.110.120717. PMC 2942870. PMID 20660643. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=2942870.
- ↑ Li T, Huang S, Jiang WZ, Wright D, Spalding MH, Weeks DP, Yang B (August 2010). "TAL nucleases (TALNs): hybrid proteins composed of TAL effectors and FokI DNA-cleavage domain". Nucleic Acids Res. DOI:10.1093/nar/gkq704. PMID 20699274.
- ↑ Smith HO, Nathans D (December 1973). "Letter: A suggested nomenclature for bacterial host modification and restriction systems and their enzymes". J. Mol. Biol. 81 (3): 419–23. DOI:10.1016/0022-2836(73)90152-6. PMID 4588280.
- ↑ Roberts RJ, Belfort M, Bestor T, Bhagwat AS, Bickle TA, Bitinaite J, Blumenthal RM, Degtyarev SKh, Dryden DT, Dybvig K, Firman K, Gromova ES, Gumport RI, Halford SE, Hattman S, Heitman J, Hornby DP, Janulaitis A, Jeltsch A, Josephsen J, Kiss A, Klaenhammer TR, Kobayashi I, Kong H, Krüger DH, Lacks S, Marinus MG, Miyahara M, Morgan RD, Murray NE, Nagaraja V, Piekarowicz A, Pingoud A, Raleigh E, Rao DN, Reich N, Repin VE, Selker EU, Shaw PC, Stein DC, Stoddard BL, Szybalski W, Trautner TA, Van Etten JL, Vitor JM, Wilson GG, Xu SY (April 2003). "A nomenclature for restriction enzymes, DNA methyltransferases, homing endonucleases and their genes". Nucleic Acids Res. 31 (7): 1805–12. DOI:10.1093/nar/gkg274. PMC 152790. PMID 12654995. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=152790.
- ↑ Geerlof A. "Cloning using restriction enzymes". European Molecular Biology Laboratory - Hamburg. 25 Mayıs 2009 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20090525040710/http://www.embl-hamburg.de:80/~geerlof/webPP/genetoprotein/cloning_strategy/clo_rest-enzymes.html. Erişim tarihi: 2008-06-07.
- ↑ Russell, David W.; Sambrook, Joseph (2001). Molecular cloning: a laboratory manual. Cold Spring Harbor, N.Y: Cold Spring Harbor Laboratory. ISBN 0-87969-576-5.
- ↑ Wolff JN, Gemmell NJ (February 2008). "Combining allele-specific fluorescent probes and restriction assay in real-time PCR to achieve SNP scoring beyond allele ratios of 1:1000". BioTechniques 44 (2): 193–4, 196, 199. DOI:10.2144/000112719. PMID 18330346.
- ↑ Zhang R, Zhu Z, Zhu H, Nguyen T, Yao F, Xia K, Liang D, Liu C (July 2005). "SNP Cutter: a comprehensive tool for SNP PCR-RFLP assay design". Nucleic Acids Res. 33 (Web Server issue): W489–92. DOI:10.1093/nar/gki358. PMC 1160119. PMID 15980518. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=1160119.
- ↑ Stryer, Lubert; Berg, Jeremy Mark; Tymoczko, John L. (2002). Biochemistry. San Francisco: W.H. Freeman. s. 122. ISBN 0-7167-4684-0.
- ↑ Roberts RJ (January 1980). "Restriction and modification enzymes and their recognition sequences". Nucleic Acids Res. 8 (1): r63–r80. DOI:10.1093/nar/8.1.197-d. PMC 327257. PMID 6243774. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pmcentrez&artid=327257.
- ↑ R.J Roberts, 1988, Nucl Acids Res. 16(suppl):271 From p.213 Molecular Cell Biology 4th Edition by Lodish, Berk, Zipursky, Matsudaira, Baltimore and Darnell.
- 1 2 3 4 5 6 7 Monty Krieger; Matthew P Scott; Matsudaira, Paul T.; Lodish, Harvey F.; Darnell, James E.; Lawrence Zipursky; Kaiser, Chris; Arnold Berk (2004). Molecular Cell Biology. New York: W.H. Freeman and Company. ISBN 0-7167-4366-3.
- ↑ "Stu I from Streptomyces tubercidicus". Sigma-Aldrich. http://www.sigmaaldrich.com/catalog/search/ProductDetail/SIGMA/R8013. Erişim tarihi: 2008-06-07.
- ↑ Shimotsu H, Takahashi H, Saito H (November 1980). "A new site-specific endonuclease StuI from Streptomyces tubercidicus". Gene 11 (3-4): 219–25. DOI:10.1016/0378-1119(80)90062-1. PMID 6260571.
Dış bağlantılar
Genel:
- Firman K (2007-11-24). "Type I Restriction-Modification". University of Portsmouth. 22 Nisan 2016 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20160422100733/http://www.typei-rm.info/. Erişim tarihi: 2008-06-06.
- Goodsell DS (2000-08-01). "Restriction Enzymes". Molecule of the Month. RCSB Protein Data Bank. 5 Temmuz 2009 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20090705070648/http://www.rcsb.org/pdb/static.do?p=education_discussion/molecule_of_the_month/pdb8_1.html. Erişim tarihi: 2008-06-06.
- Simmer M, Secko D (2003-08-01). "Restriction Endonucleases: Molecular Scissors for Specific2ally Cutting DNA". The Science Creative Quarterly. 11 Ekim 2007 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20071011132838/http://scq.ubc.ca:80/?p=249. Erişim tarihi: 2008-06-06.
Veritabanları:
- Roberts RJ, Vincze T, Posfai, J, Macelis D. "REBASE". http://rebase.neb.com. Erişim tarihi: 2008-06-06. "Restriction Enzyme Database"
Yazılım:
- Bikandi J, San Millán R, Rementeria A, and Garaizar J. "Restriction enzyme digest of DNA". insilico.ehu.es. 27 Mayıs 2016 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20160527001046/http://insilico.ehu.es/restriction/. Erişim tarihi: 2008-06-06.
- Palmer M. "WatCut". University of Waterloo, Ontario, Canada. 18 Haziran 2007 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20070618114401/http://watcut.uwaterloo.ca:80/watcut/watcut/template.php. Erişim tarihi: 2008-06-06. "An on-line tool for restriction analysis, silent mutation scanning, SNP-RFLP analysis"
- Vincze,T, Posfai J, Roberts RJ. "NEBcutter V2.0". New England Biolabs Inc.. 19 Ağustos 2010 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20100819190538/http://tools.neb.com/NEBcutter2/index.php. Erişim tarihi: 2008-06-06. "Restriction enzyme finder"
- "Restriction enzyme digest of DNA software". BioPHP: PHP for Bioinformatics. 13 Mart 2016 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20160313025142/http://biophp.org/minitools/restriction_digest/demo.php. Erişim tarihi: 2008-06-06. "Online tool, free source code"
- "pDRAW32". AcaClone software. 22 Nisan 2016 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20160422072652/http://www.acaclone.com/. Erişim tarihi: 2008-06-06. "Freeware DNA cloning, sequence analysis and plasmid/DNA plotting software"