DNA
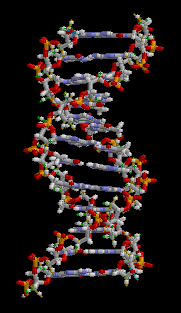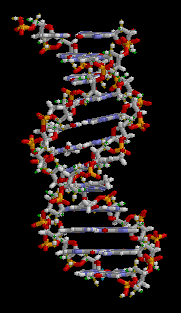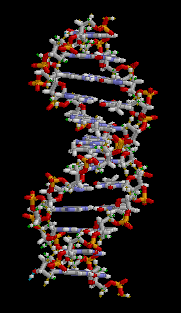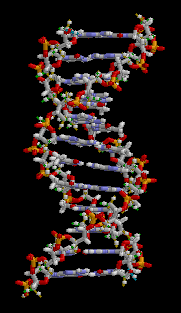
Deoksiribo Nükleik asit veya kısaca DNA, tüm organizmalar ve bazı virüslerin canlılık işlevleri ve biyolojik gelişmeleri için gerekli olan genetik talimatları taşıyan bir nükleik asittir. DNA'nın başlıca rolü bilginin uzun süreli saklanmasıdır. Protein ve RNA gibi hücrenin diğer bileşenlerinin inşası için gerekli olan bilgileri içermesinden dolayı DNA; bir kalıp, şablon veya reçeteye benzetilir. Bu genetik bilgileri içeren DNA parçaları gen olarak adlandırılır. Ama başka DNA dizilerinin yapısal işlevleri vardır (kromozomların şeklini belirlemek gibi), diğerleri ise bu genetik bilginin ne şekilde (hangi hücrelerde, hangi şartlarda) kullanılacağının düzenlenmesine yararlar.
Kimyasal olarak DNA, nükleotit olarak adlandırılan basit birimlerden oluşan iki uzun polimerden oluşur. Bu polimerlerin omurgaları, ester bağları ile birbirine bağlanmış şeker ve fosfat gruplarından meydana gelir. Bu iki iplik birbirlerine ters yönde uzanırlar. Her bir şeker grubuna baz olarak adlandırılan dört tip molekülden biri bağlıdır. DNA'nın omurgası boyunca bu bazların oluşturduğu dizi, genetik bilgiyi kodlar. Protein sentezi sırasında bu bilgi, genetik kod aracılığıyla okununca proteinlerin amino asit dizisini belirler. Bu süreç sırasında DNA'daki bilgi, DNA'ya benzer yapıya sahip başka bir nükleik asit olan RNA'ya kopyalanır. Bu işleme transkripsiyon denir.
Hücrelerde DNA, kromozom olarak adlandırılan yapıların içinde yer alır. Hücre bölünmesinden evvel kromozomlar eşlenir, bu sırada DNA ikileşmesi gerçekleşir. Ökaryot canlılar (yani hayvan, bitki, mantar ve protistalar) DNA'larını hücre çekirdeği içinde bulundururken prokaryot canlılarda (yani bakteri ve arkelerde) DNA, hücre sitoplazmasında yer alır. Kromozomlarda bulunan kromatin proteinleri (histonlar gibi) DNA'yı sıkıştırıp organize ederler. Bu sıkışık yapılar DNA ile diğer proteinler arasındaki etkileşimleri düzenleyerek DNA'nın hangi kısımlarının okunacağını kontrol eder.
Özellikler

Nükleotit olarak adlandırılan birimlerden oluşan bir polimerdir.[1][2] DNA zinciri 22 ila 26 Ångström arası (2,2-2,6 nanometre) genişliktedir, bir nükleotit birim 3,3 Å (0.33 nm) uzunluğundadır.[3] Her bir birim çok küçük olmasına rağmen, DNA polimerleri milyonlarca nükleotitten oluşan muazzam moleküllerdir. Örneğin, en büyük insan kromozomu olan 1 numaralı kromozom yaklaşık 220 milyon baz çifti uzunluğundadır.[4]
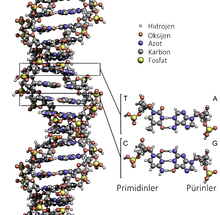
Dna'nın yarısı dişi bireyden yarısı da erkek bireyden gelir. Canlılarda DNA genelde tek bir molekül değil, birbirine sıkıca sarılı bir çift molekülden oluşur.[5][6] Bu iki uzun iplik sarmaşık gibi birbirine sarılarak bir çift sarmal oluşturur. Nükleotit birimler bir şeker, bir fosfat ve bir bazdan oluşurlar. Şeker ve fosfat DNA molekülünün omurgasını oluşturur, baz ise çifte sarmaldaki öbür DNA ipliği ile etkileşir. Genel olarak bir şekere bağlı baza nükleozit, bir şeker ve bir veya daha çok fosfata bağlı baza ise nükleotit denir. Birden çok nükleotidin birbirine bağlı haline polinükleotit denir.[7]
DNA ipliğinin omurgası almaşıklı şeker ve fosfat artıklarından oluşur.[8] DNA'da bulunan şeker 2-deoksiribozdur, bu bir pentozdur (beş karbonlu şekerdir). Bitişik iki şekerden birinin 3 numaralı karbonu ile öbürünün 5 numaralı karbon atomu arasındaki fosfat grubu, bir fosfodiester bağı oluşturarak şekerleri birbirine bağlar. Fosfodiester bağın asimetrik olması nedeniyle DNA ipliğinin bir yönü vardır. Çifte sarmalda bir iplikteki nükleotitlerin birbirine bağlanma yönü, öbür ipliktekilerin yönünün tersidir. DNA ipliklerinin bu düzenine anti-paralel denir. DNA ipliklerin asimetrik olan uçları 5' (beş üssü) ve 3' (üç üssü) olarak adlandırılır, 5' uç bir fosfat grubu, 3' uç ise bir hidroksil grubu taşır. DNA ve RNA arasındaki başlıca farklardan biri, içerdikleri şekerdir, RNA'da 2-deoksiriboz yerine başka bir pentoz şeker olan riboz bulunur.[6]
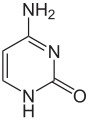
Çift sarmalı iki ipliğe bağlı bazlar arasındaki hidrojen bağları DNA'yı stabilize eder. DNA'a bulunan dört baz, adenin (A olarak kısaltılır), sitozin (C), guanin (G) ve timin (T) olarak adlandırılır. Bu dört baz şeker-fosfata bağlanarak bir nükleotit oluşturur, örneğin "adenozin monofosfat" bir nükleotittir.
Bazlar iki tip olarak sınıflandırılırlar: adenin ve guanin, pürin türevleridir, bunlar beş ve altı üyeli halkaların kaynaşmasından oluşmuş heterosiklik bileşiklerdir; sitozin ve timin ise pirimidin türevleridir, bunlar altı üyeli bir halkadan oluşur. Bir diğer baz olan urasil (U), sitozinin yıkımı sonucu seyrek olarak DNA'da bulunabilir. Kimyasal olarak DNA'ya benzeyen RNA'da timin yerine urasil bulunur.
Oyuklar

İki sarmal iplik DNA omurgasını oluşturur. Bu iplikler araındaki boşluklar takip edilerek iki tane hayali boşluk veya oyuk daha bulunabilir. Bu oyuklar baz çiftlerine bitişiktir ve onlara bağlanmak için bir yer oluşturabilirler. Bu oyuklar birbirlerinin tam karşısında olmadıkları için büyüklükleri aynı değildir. Bunlardan büyük oyuk (majör oyuk) olarak adlandırılanı 22 Å genişliğinde, küçük (minör) oyuk ise 12 Å genişliğindedir.[9] Küçük oyuğun darlığı nedeniyle bazların kenarlarına erişmek büyük oyuktan daha kolaydır. Bu nedenle, DNA'daki belli baz dizilerine bağlanan, transkripsiyon faktörü gibi proteinler büyük oyuktan bazların kenarlarına temas ederler.[10] Hücredeki DNA'nın bazı bölgelerinde bu durum farklı olabilir (aşağıda "Alternatif çifte sarmal yapılar" bölümüne bakınız) ama oralarda dahi, eğer DNA normal B biçimini alacak şekilde burulsaydı görülecek büyüklük farklılıklarına göre adlandırılır.
Baz eşleşmesi
- Daha fazla bilgi: Baz çifti
 |
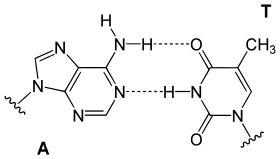 |
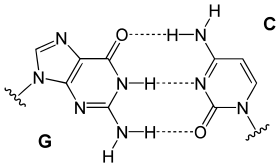
DNA'nın bir ipliğindeki bir baz tipi, öbür iplikten tek bir baz tipi ile bağ kurar. Buna tümleyici (komplemanter) baz eşleşmesi denir: pürinler pirimidinler ile hidrojen bağı kurar, A yalnızca T'ye bağlanır, C'de yalnızca G'ye bağlanır. Çift sarmalda karşıdan karşıya birine bağlı iki baza bir baz çifti denir. Çift sarmalı kararlı kılan ayrıca hidrofobik etki ve pi istiflenmesi vardır, bunlar DNA dizisisinden bağımsızdır.[11] Hidrojen bağları kovalent bağlardan daha zayıf olduklarından kolayca kopup tekrar oluşabilirler. Dolayısıyla DNA zincirinin iki ipliği bir fermuar gibi kolayca birbirinden ayrılabilir, ya mekanik güç ile veya yüksek sıcaklıkta.[12] Komplementerliğin bir sonucu olarak bir DNA sarmalındaki iki iplikli dizideki tüm bilgi ipliklerin her birinde kopyalanmış durumdadır, bu da DNA kopyalanması için esas bir özelliktir. Aslında komplementer baz çiftleri arasındaki spesifik ve tersinir etkileşimler DNA'nın canlılardaki işlevleri için şarttır.[1]
İki tip baz çifti farklı sayıda hidrojen bağları oluşturur, AT'nin iki hidrojen bağı, GC'nin üç hidrojen bağı vardır (bakınız şekil). Dolayısıyla GC çiftleri AT baz çiftlerinden daha güçlüdür. Dolayısyla iki DNA ipliğinin birbirine bağlanma gücünü belirleyen, hem DNA çift sarmalının uzunluğu hem de onu oluşturan GC baz çiftlerinin yüzde oranıdır. Yüksek oranda GC'li uzun DNA'ların iplikleri birbirine daha sıkı bağlıdır, AT oranı yüksek kısa sarmalların iplikleri ise birbiriyle daha zayıf etkileşirler.[13] Biyolojide, DNA çifte sarmalının kolay ayrılması gereken bölgelerinde AT oranı yüksek olur, örneğin bazı promotörlerde bulunan TATAAT Pribnow kutusu.[14] Laboratuvarda bu etkileşimin gücünü ölçmek için hidrojen bağlarını koparmak için gerekli sıcaklık, ergime sıcaklığı belirlenir (bu, Tm sıcaklığı olarak da adlandırılır). DNA çifte sarmalındaki tüm baz çiftleri eridikten sonra iplikler ayrışır ve çözeltide iki bağımsız molekül olarak varlığını sürdürür. Bu iki tek iplikli DNA molekülün tek bir biçimi yoktur, ama bazı biçimler diğerlerinden daha kararlıdır.[15]
Anlam ve ters anlam
- Daha fazla bilgi: Anlam (moleküler biyoloji)
Bir DNA dizisi, eğer ondan protein sentezlemeye yarayan mesajcı RNA kopyası ile aynı diziye sahipse, "anlamlı" olduğu söylenir.[16] Öbür iplikteki diziye "ters anlamlı" dizi denir. Aynı DNA ipliğinin farklı bölgelerinde anlamlı ve ters anlamlı diziler bulunabilir, yani her iki iplikte hem anlamlı hem anlamsız diziler bulunur. Hem prokaryot ve ökaryotlarda ters anlamlı, yani protein üretimine yaramayan, RNA'nın üretildiği olur, bu RNA'ların işlevi halen tam bilinmemektedir.[17] Bir görüşe göre ters anlamlı RNA, RNA-RNA baz eşleşmesi yoluyla gen ifadesinin düzenlenmesine yaramaktadır.[18]
Bazı DNA dizilerinde anlam ve ters anlam kavramları birbirine karışır, çünkü bazen genler birbiriye örtüşebilir.[19] Böyle durumlarda bazı DNA dizileri çifte görev yapar, bir iplik boyunca okununca bir protein kodlar, öbür iplik boyunca okununca ikinci bir protein kodlar. Bakterilerde bu tür gen örtüşmeleri gen transkripsiyonunun düzenlenmesi ile ilişkili olduğuna dair bulgular vardır,[20] virüslerde ise, genlerin örtüşmesi küçük bir viral genoma daha çok bilginin sığmasını sağlar.[21]
Süper burulma
- Daha fazla bilgi: DNA süpersarımı
Süper burulma (İngilizce supercoiling) tabir edilen bir süreç ile DNA bir halat gibi burulabilir. "Gevşek" halinde DNA'daki bir iplik, her 10,4 baz çiftinde bir, çift sarmalın ekseni etrafında bir tam dönüş yapar. Ama, eğer DNA burulursa iplikler daha sıkı veya daha gevşek sarılı olabilirler.[22] Eğer DNA sarmalı sarılma yönünde burulursa buna pozitif süperburulma denir ve bazlar birbirlerine daha sıkı şekilde tutunurlar. Eğer ters yönde burulursa DNA, buna negatif süperburulma denir ve bazlar birbirlerinden daha kolay ayrışırlar. Doğadaki çoğu DNA molekülü az derecede negatif süper burguludur, bundan topoizomeraz adlı enzimler sorumludur.[23] Bu enzimlerin bir işlevi transkripsiyon ve DNA ikileşmesi gibi süreçler sırasında DNA ipliklerine etki eden burulmayı bertaraf etmektir.[24]
Alternatif çifte sarmal yapılar

- Daha fazla bilgi: DNA yapısı
DNA'nın çeşitli biçimleri (konformasyonları) mevcuttur.[8] Ancak, canlılarda sadece A-DNA, B-DNA, ve Z-DNA gözlemlenmiştir. DNA'nın hangi biçimi aldığı DNA dizisine, süperburulmanın yönü ve miktarına, bazlardaki kimyasal değişimlere, ve çözeltinin özelliklerine (metal iyonu ve poliamin konsantrasyonu gibi) bağlıdır.[25] Bu üç biçimden yukarıda betimlenmiş olan "B" biçimi, hücrelerde bulunan şartlar altında en sık görülenidir.[26]
B biçimine kıyasla DNA'nın A biçimi daha geniş bir sarmaldır, küçük oluk daha geniş ve sığ, büyük oluk da daha dar ve derindir. A biçimli nükleik asitler, fizyolojik olmayan şartlarda, suyunu kaybetmiş DNA örneklerinde görülür, hücre içinde ise DNA ve RNA ipliklerinin birbirine sarılmasından oluşan karma (hibrit) eşleşmelerde, ayrıca bazı enzim-DNA komplekslerinde meydana gelebilir.[27][28] Metilasyonla kimyasal değişime uğrayan DNA parçaları daha büyük biçimsel değişiklik gösterip Z biçimini alabilirler. Bu durumda iplikler sarmal ekseni etrafında dönerek sol elli bir spiral oluşturur, bu daha yaygın olan B biçimimdekinin tersi yöndedir.[29] Bu sıra dışı yapılar Z-DNA bağlayıcı proteinler tarafından tanınır ve transkripsiyon kontrolü ile ilişkili olduğu sanılmaktadır.[30]
Dörtlü Sarmal yapılar
- Daha fazla bilgi: G-dörtlüsü
Doğrusal kromozomların uçlarında telomer olarak adlandırılan özelleşmiş bölgeler bulunur. Bu bölgelerin ana fonksiyonu kromozom uçlarının telomeraz adlı enzim aracılığıyla kopyalanmasını sağlamaktır. DNA'yı normalde kopyalayan enzimler kromozomların en uç kısımların kopyalayamadığı için bu kopyalama telomeraz aracılığıyla yapılır.[32] Bu özelleşmiş kromozom başlıkları ayrıca DNA'nın uçlarını korurlar ve hücredeki DNA tamir sistemlerinin bunları tamir edilmesi gereken hasar olarak algılanmasını engeller.[33] İnsan hücrelerinde telomerler genelde TTAGGG dizisinin birkaç bin kere tekrarından oluşan tek iplikli DNA uzantılarıdır.[34]
Bu guanin zengini diziler normal DNA'daki baz çiftleri yerine, dört bazlı birimlerden meydana gelmiş istiflenme kümeleri ile kromozom uçlarını stabilize ederler. Burada dört guanin bazı yassı bir tabaka oluştururlar, bunlar da birbiri üzerine istiflenerek kararlı bir G-dörtlüsü (G-quadruplex) yapısı oluştururlar.[35] Bu yapıların stabilizasyonu, bazların kenarları arasındaki hidrojen bağları ve her dört bazlı birimin ortasında yer alan bir metal iyonun şelasyonu ile gerçekleşir.[36] Bu G-dörtlüleri başka yollardan da oluşabilir: tek bir ipliğin birkaç kere katlanması ile bu dörtlü birim oluşabilir, veya ikiden fazla farklı paralel ipliğin her birinin ortak yapıya bir baz temin etmesi ile de bu dört baz bir araya gelebilir.
Bu istiflenmiş yapıların aynı sıra, telomerler ayrıca telomer ilmiği (T-ilmiği; İngilizce: telomere loops veya T-loops) adlı yapılar oluştururlar. Bunlarda tek iplikli DNA, telomer bağlanıcı proteinler tarafından stabilize edilmiş bir halka olarak kıvrılır.[37] Bir T-ilmiğinin en ucundaki tek iplikli DNA, çift iplikli bir DNA bölgesine bağlıdır. Bu birleşme noktasında tek iplikli telomer DNA'sı, çift iplikli DNA'nın çifte sarmalını bozup iki sarmaldan biri ile baz eşleşmesi yapar. Bu üç sarmallı yapıya yer değişim halkası (İngilizce displacement loop veya D-loop) denir.[35]
Kimyasal değişimler
 |
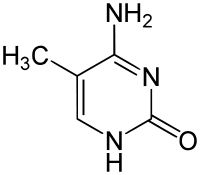 |
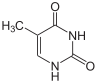 |
| sitozin | 5-metilsitozin | timin |
Baz değişimleri
- Daha fazla bilgi: DNA metilasyonu
Kromatin adı verilen bir yapı içinde DNA'nın paketlenmesi ile kromozomlar meydana gelir. Bu paketlenme gen ifadesine etki eder. Baz değişimi (modifikasyonu) bu paketlenmeyle ilişkilidir, öyle ki gen ifadesinin az olduğu veya hiç olmadığı yerlerde sitozin bazları yüksek derecede metilasyona uğramıştır. Örneğin, sitozin metilasyonu ile 5-metilsitozin meydana gelir, bu X kromozomu inaktivasyonu için önemlidir.[38] Ortalama metilasyon düzeyi canlıdan canlıya farkeder: solucan Caenorhabditis elegans'da sitozin metilasyonu olmaz, buna karşın omurgalı DNA'sının %1'e ulaşan kadarı 5-metilsitozin içerebilir.[39] 5-metilsitozinin önemli bir baz olmasına rağmen, onun deaminasyonu sonucu bir timin bazı oluşur, bu yüzden metillenmiş sitozinler mutasyona eğilimlidirler.[40] Diğer baz modifikasyonarı arasında bakterilerde görülen adenin metilasyonu ve kinetoplastitlerde urasilin glikozilasyonu sonunda meydana gelen "J-bazı" sayılabilir.[41][42]
DNA hasarı
- Daha fazla bilgi: Mutasyon
DNA çeşitli farklı mutajenler tarafından hasara uğrayabilir, bunun sonucunda DNA dizisi değişebilir. Mutajenler arasında başlıca, yükseltgen (oksitleyici) etmenler, alkilleyici etmenler ve yüksek enerjili elektromanyetik ışınlar (morötesi ışık ve X ışınları gibi) sayılabilir. DNA'da meydana gelen hasarın tipi mutagenin tipine bağlıdır. Örneğin, mor ötesi ışık timin ikilileri (timin dimerleri) oluşturarak DNA'ya hasar verir.[44] Buna karşın, serbest radikaller veya hidrojen peroksit gibi yükseltgen etmenler çeşitli farklı türden hasar oluşturabilirler, baz değişimi (özellikle guanozin) ve iki iplikli kırılmalar gibi.[45] Her bir insan hücresinde günde 500 baz yükseltgeyici zarar görür.[46][47] Bu yükseltgeyici hasarlardan en zararlısı çift zincirli kırılmalardır, çünkü bunların onarımı zordur, bunlar DNA dizilerinde noktasal mutasyonlara, insersiyonlara ve delesyonlara ayrıca kromozomal translokasyonlara yol açabilirler.[48]
Çoğu mutajen, iki baz çifti arasındaki boşluğa girer, buna enterkalasyon denir. Çoğu enterkalatörler aromatik ve düzlemsel moleküllerdir, bunlara örnek olarak etidyum bromür, daunomisin ve doksorubisin sayılabilir. Bir enterkalatörün iki baz çifti arasına girebilmesi için bunların arasının açılması, bunun olabilesi için de DNA sarmalının normalin aksi yönde burularak gevşemesi gerekir. Bunlar olunca transkripsiyon ve DNA ikilenmesi engellenir, zehirlenme ve mutasyonlar meydana gelir. Bu yüzden DNA enterkalatörleri çoğunlukla kanserojendir, bunların iyi bilinen örnekleri olarak benzopiren diol epoksit, akridin türevleri aflatoksin ve etidyum bromür sayılabilir.[49][50][51] Tüm bunlara rağmen, DNA transkripsiyonuna engel olma özelliklerinden dolayı bu toksinler aynı zamanda hızla büyüyen kanser hücrelerini engellemek amacıyla kemoterapide kullanılırlar.[52]
Biyolojik işlevleri
DNA, ökaryotlarda doğrusal kromozomlar, prokaryotlarda ise dairesel kromozomlar içinde bulunur. Bir hücredeki kromozomlar kümesine onun genomu denir; insan genomu 46 kromozom içinde yer alan yaklaşık 3 milyar baz çiftinden oluşur.[53] Protein ve diğer işlevsel RNA molekülleri kodlayan bilgi, gen adı verilen DNA parçalarının dizisinde yer alır. Genlerdeki genetik bilginin aktarılması baz eşleşmesi ile gerçekleşir. Örneğin, transkripsiyon sırasında bir DNA dizisinin ona komplementer bir RNA dizisi olarak kopyalanması, DNA ile doğru RNA nükleotitler arasındaki çekim ile mümkün olur. Protein çevrimi (translasyon) denen süreç sırasında bu RNA dizisine kaşılık gelen bir protein sentezlenirken, RNA nükleotitleri arasında gene baz eşleşmesi olur. Bir diğer önemli biyolojik süreç, hücredeki genetik bilginin kopyalanması olan DNA ikilenmesidir. Bu işlevlerin ayrıntıları başka maddelerde işlenmiştir; burada DNA ile genomun fonksiyonlarını yerine getiren diğer moleküller arasındaki etkileşimler ele alınmıştır.
Genler ve genomlar
- Daha fazla bilgi: Hücre çekirdeği, Kromatin, Kromozom, Gen, kodlamayan DNA
Genomu oluşturan DNA ökaryotlarda hücre çekirdeğinde, ayrıca az miktarda mitokondrilerde bulunur. Prokaryotlardaki DNA, sitoplazma içinde yer alan, düzensiz şekilli nükleoit denen cismin içindedir.[54] Genom tarafından kodlanan bilgi genlerde yer alır, bir canlı birey tarafından taşınan bu bilginin tamamına onun genotipi denir. Gen kalıtımsal bir birimdir ve organizmanın belli bir özelliğini belirleyen bir DNA dizisi ile tanımlanır. Ayrıca, bu DNA bölgesinin transkripsiyonunu düzenleyen diziler (promotör ve hızlandırıcılar gibi) de vardır.
Çoğu biyolojik türde genomdaki dizilerin ancak ufak bir bölümü protein kodlar. Örneğin insan genomunun ancak %1'i protein eksonları kodlar, buna karşın insan DNA'sının %50'si protein kodlamayan, kendini tekrar eden dizilerden oluşur.[55] Ökaryot genomlarında bu kadar çok protein kodlamayan DNA'nın bulunması ve türlerin genom büyüklüğündeki ("C-değeri"ndeki) büyük farklılıkların nedeni henüz anlaşılamamıştır ve "C değeri muamması" olarak bilinir.[56] Ancak, protein kodlamayan (non-coding) DNA dizileri gene de işlevsel kodlamayan RNA molekülleri kodlamaktadır, bunlar da gen ifadesinin düzenlenmesinde rol oynarlar.[57]
Bazı kodlamayan DNA dizileri kromozomlar için yapısal rol oynarlar. Telomer ve sentromerler tipik olarak çok az sayıda gen içerir, ama kromozomların işlev ve stabilitesi için önemlidir.[33][59] İnsanlarda bulunan kodlamayan DNA'ların önemli bir türü psödogenlerdir, bunlar mutasyon sonucu çalışmaz hale gelmiş genlerin kopyalarıdır.[60] Bu DNA dizileri genelde birer moleküler fosilden ibarettir ama bazen yeni genlerin oluşumuna ham madde olabilirler, gen ikilenmesi ve ıraksak evrim süreçleri sonucu.[61]
Transkripsiyon ve çevrim
- Daha fazla bilgi: Genetik kod, Transkripsiyon (genetik), Protein biyosentezi
Genler, işlevsel moleküller kodlayan DNA dizileridir, bunlar canlının fenotipini belirler. Protein kodlayan genler durumunda DNA dizisi bir mesajcı RNA dizisini tanımlar, bu da bir veya birkaç proteinin dizisini belirler. Genlerdeki DNA dizisi ile proteinlerdeki amino asit dizisi arasındaki ilişki, biyolojik çevrim (translasyon) kuralları tarafından belirlenir, bunlar topluca genetik kod ile özetlenir. Genetik kod, üç nükleotitlik dizilere karşılık gelen, üç harfli 'kelimelerden' oluşur (örneğin, ACT, CAG, TTT), bu üçlüler kodon olarak adlandırılır.
Transkripsiyonda, protein kodlayan bir genin kodonları önce RNA polimeraz tarafından bir mesajcı RNA şeklinde kopyalanır. Bu RNA kopya, ardından bir ribozom tarafından deşifre edilir; ribozom, mesajcı RNA ile amino asit taşıyan taşıyıcı RNA'lar arasında baz eşlemesi yaparak onu okur. Dört bazın 3'lü kombinasyonları olabildiği için 64 olası kodon vardır ( kombinasyon). Bunlar yirmi standart amino asidi kodlarlar, böylece çoğu amino asite birden çok kodon düşer. Ayrıca, protein kodlayıcı bölgenin sonuna işaret eden üç tane de 'stop' veya anlamsız (nonsense) kodon vardır, bunlar TAA, TGA ve TAG kodonlarıdır.
İkileşme
- Daha fazla bilgi: DNA ikileşmesi

Canlıların çoğalması ve (çok hücreli canlıların) büyümesi için hücre bölünmesi gereklidir. Ancak bir hücre bölünürken DNA'sını da kopyalamak zorundadır ki iki yavru hücre ana hücredeki genetik bilginin aynısına sahip olsunlar. DNA'nın iki iplikli yapısı DNA ikileşmesi (DNA duplikasyonu) için basit bir mekanizma sağlar. İki iplik ayrışırlar, sonra her bir iplikteki dizinin komplementer dizisi DNA polimeraz adlı bir enzim tarafından imal edilir. Bu enzim, tümleyici ipliği sentezlemek için gereken her bazın doğru olanını baz eşleşmesi yoluyla seçer ve onu uzamakta olan ipliğe ekler. DNA polimeraz bir DNA ipliğini ancak 5' - 3' yönünde uzatabildiği için, bir çifte sarmalın antiparalel ipliklerininin kopyalanması için farklı mekanizmalar mevcuttur.[62] Böylece, eski iplikteki baz, yeni ipliğe eklenen bazları belirler, sonunda hücre DNA'sının mükemmel bir kopyasını elde eder.
Proteinler ile etkileşim
DNA'nın tüm işlevleri onun proteinlerle olan etkileşimine bağlıdır. Bu protein etkileşimlerinin bazıları özgül-dışıdır (non-spesifiktir), bazılarında ise protein ancak belli bir DNA dizisine bağlanabilir. Enzimler de DNA'ya bağlanabilir ve bunlar arasında DNA baz disini transkripsiyon ve DNA ikilemesi için kopyalayan polimerazlar özellikle çok önemlidir.
DNA'ya bağlanıcı proteinler
 |
DNA'ya bağlanan yapısal proteinler, non-spesifik DNA-protein etkileşimlerinin iyi anlaşılmış örneklerindendir. Kromozomlarda bulunan DNA, yapısal proteinlerle beraber kompleksler oluşturur. Bu proteinler DNA'yı kromatin adlı kompakt yapı içinde organize ederler. Ökaryotlarda kromatinin oluşmasında DNA'nın histon adlı küçük, bazik proteinlere bağlanması önemli bir rol oynar; prokaryotlarda ise çeşitli başka protein türleri DNA'ya bağlanır.[63][64] Histonlar, nükleozom adlı disk şeklinde bir kompleks oluştururlar, çift iplikli DNA buna sarılarak iki kere bunun etrafında döner. Histonların bazik kalıntıları ile DNA'nın şeker-fosfat omurgasındaki asidik fosfatlar arasındaki iyonik bağlar, non-spesifik bir etkileşim oluşturur, baz dizisinden büyük ölçüde bağımsızdırlar.[65] Bu bazik amino asitlerin kimyasal değişimleri arasında metilasyon, fosforilasyon, ve asetilasyon sayılabilir.[66] Bu kimyasal değişimler, DNA'nın histonlarla etkileşimini etkiler, bunun sonucunda DNA'ya transkripsyon faktörlerinin erişimi ve transkripsiyon hızı değişir.[67] Kromatinde bulunan diğer non-spesifik DNA'ya bağlanıcı proteinler arasında bulunan yüksek hareketli grup proteinleri (ing. high-mobility group proteins) bükülmüş veya distorte olmuş DNA'ya bağlanır.[68] Bu proteinler, bitişik nükleozom gruplarını bükerek daha büyük ölçekli yapılar oluşturarlar ve kromozomları meydana getirirler.[69]
DNA'ya bağlanıcı proteinler arasında bulunan başlıca bir protein grubu, tek iplikli DNA'ya bağlanıcı proteinlerdir (bunlar tek iplikli DNA bağlayıcı protein olarak da adlandırılırlar). İnsanda replikasyon protein A bu protein ailesinin en iyi anlaşılmış üyesi sayılır, bu protein, cifte sarmalın ayrıştığı durumlarda, örneğin DNA ikileşmesi, rekombinasyon ve DNA tamirinde işlev görür.[70] Bu proteinler tek iplikli DNA'yı kararlı kılar, onun sap-ilmik (stem-loop) oluşturmasına veya nükleazlar tarafında yıkımına engel olurlar.

Yukarıda değinilen proteinlerden farklı olarak başka proteinler belli DNA dizilerine bağlanacak şekilde evrimleşmişlerdir. Bunların en iyi araştırılmış olanları transkripsiyon faktörleridir, bular transkripsiyonu düzenleyen proteinlerdir. Her transkripsiyon faktörü belli bir DNA diziler kümesine bağlanır ve bu dizilere yakın protörleri olan genlerin transkripsiyonu etkinleştirir veya engeller. Transkripsiyon faktörleri bunu iki farklı yoldan gerçekletirir. Birincisi, transkripsiyondan sorumlu olan RNA polimeraz bağlanırlar, bunu ya doğrudan ya da aracı proteinlerle yaparlar, bunun sonucunda polimeraz promotöre yakın bir konuma yerleştitilmiş olur ve transkripsiyona başlaması mümkün hale gelir.[72] Bir diğer yolda ise, transkripsiyon faktörleri promotörde yer alan histonları kimyasal değişime uğratan enzimlere bağlanırlar; bunun sonucunda polimerazın DNA'ya erişimi değişir.[73]
Bu DNA bağlanma dizileri bir canlının genomunun her tarafında bulunabileceği için, bir transkripsiyon faktörünün etkinliğinde meydan gelen değişiklikler binlerce gene etki edebilir.[74] Dolayısıyla bu proteinler çoklukla, çevresel değişiklikler, hücresel başkalaşım ve gelişimi kontrol eden süreçlerle ilişkili olan sinyal iletim süreçlerinin hedefidirler. Bu transkripsiyon faktörlerinin DNA ile etkileşimindeki spesifisite, proteinin DNA bazlarının kenarları ile yaptığı temaslardan kaynaklanmaktadır, bu sayede bu proteinler DNA'nın dizisini "okurlar". Bazlarla olan bu etkileşimlerin çoğu, bu bazlara kolaylıkla erişilebilen büyük olukta meydan gelir.[75]

DNA değiştirici enzimler
Nükleaz ve ligazlar
Nükleazlar DNA iplikleri kesen enzimlerdir, fosfodiester bağlarının hidrolizini katalizlerler. DNA ipliklerinin uçlarındaki nükleotitleri hidrolizleyen nükleazlare eksonükleaz denir, ipliklerin iç kısımlarındaki bağları hidrolizleyenlere ise endonükleaz. Moleküler biyolojide en sık kullanılan endonükleazlar restriksiyon endonükleazlarıdır, bunlar DNA'yı belli dizilerde keserler. Örneğin soldaki resimde görülen EcoRV enzimi 6 bazlı 5'-GAT|ATC-3' dizisini tanır ve dik çizgi ile gösterilen noktada onu keser. Doğada bu enzimler, restriksiyon modifikasyon sisteminin bir parçası olarak, bakterileri fajlara karşı korumaya yararlar, hücrenin içine giren faj DNA'sını sindirerek.[77] Teknolojide bu enzimler moleküler klonlama ve DNA parmakizlemesi için kullanılır.
DNA ligaz enzimleri kesilmiş veya kırık DNA ipliklerini birleştirir.[78] Ligazlar özellikle gecikmeli iplik DNA ikileşmesinde önemli bir rol oynarlar, çünkü replikasyon çatalında meydana gelen kısa DNA parçalarını birleştirirler. Ayrıca DNA tamiri ve genetik rekombinasyonda kullanılırlar.
Topoizomeraz ve helikazlar
Topoizomerazlar hem nükleaz hem de ligaz etkinliğine sahiptir. Bu proteinler DNA'daki süperburulma derecesini değiştirirler. Bu enzimlerin bazıları DNA sarmalının bir ipliğini kesip bunun öbürü etrafında dönmesini sağlar, sonra da DNA'daki kesiği tekrar birleştirir.[23] Bu enzimlerin diğerleri ise DNA sarmalının bir ipliğini kesip öbür ipliğin bu kesiğin içinden kesmesini sağlarlar, sonra kesiği tekrar birleştirirler.[79] Topoizomerazlar DNA'yla ilgili pek çok süreçte yer alırlar, DNA ikileşmesi ve transkripsiyonu gibi.[24]
Helikazlar moleküler motor özellikli proteinlerdir. Nükleozit trifosfatlarda, özellikle ATP'de taşınan kimyasal enerjiyi kullanıp bazlar arasındaki hidrojen bağlarını kırarlar ve DNA çifte sarmalını ters yönde burarak onu tek iplikler halinde açarlar.[80] Bu enzimler DNA bazlarına erişmeye gerek duyan enzimlerin bulunduğu süreçlerde gereklidir.
Polimerazlar
Nükleik asit polimerazları, nükleozit trifosfatlardan polinükleotit zincirler sentezleyen enzimlerdir. Ürettikleri ürünler var olan polinükleotit zincirlerinin (bunlara kalıp denir) kopyalarıdır. Bu enzimler, bir DNA zincirindeki en son nükleotitin 3' hidroksil grubuna yeni bir nükleotit ekleyerek çalışır. Dolayısıyla tüm polimerazlar 5' - 3' doğrultusunda ilerler.[81] Bu enzimlerin aktif bölgesinde, gelen nükleozit trifosfat kalıp ile baz eşleşmesi yapar; bu sayede polimeraz, kalıba komplementer bir ipliği doğru bir şekilde sentezleyebilir. Polimerazlar kullandıkları kalıbın tipine göre sınıflandırılır.
DNA ikileşmesinde, DNA-bağımlısı DNA polimeraz, bir DNA dizisinin kopyasını yapar. Bu süreçte hata olmaması hayatî önem taşıdığı için bu tip polimerazlarının çoğunda prova okuma aktivitesi bulunur. Bunlarda, sentez reaksiyonunda meydana gelen ender hatalar, baz eşleşmesinin doğru olmamasıyla anlaşılır. Eğer bir uyumsuzluk algılanırsa, 3'-5' yönünde çalışan bir eksonükleaz aktivitesi etkinleştirilir ve hatalı baz çıkartılır.[82] Çoğu canlıda DNA polimerazlar replizom olarak adlandırılan ve yardımcı alt birimler (DNA kıskacı ve helikazlar gibi) içeren büyük bir kompleks içinde yer alır.[83]
RNA-bağımlısı DNA polimerazlar RNA ipliğinde bulunan diziyi DNA olarak kopyalayan özel bir polimeraz sınıfıdır. Ters transkiptazlar bu sınıfa dahildir, bunlar viral enzimler olup hücrelerin retrovirüsler tarafından enfeksiyonunda yer alırlar. Telomerazlar da bu sınıfa dahildir, bunlar da telomerlerin ikilenmesi için gereklidir.[32][84] Telomerazı diğer bu tip enzimlerden farklı kılan bir özelliği, kullandığı RNA kalbın kendi yapısının bir parçası olmasıdır.[33]
Transkripsiyon, DNA-bağımlısı RNA polimeraz tarafından gerçekleştirilir, bu enzim DNA ipliğindeki diziyi RNA olarak kopyalar. Bir genin transkripsiyonu için RNA polimeraz, DNA üzerinde promotör adlı bir bölgeye bağlanır ve DNA ipliklerini ayrıştırır. Sonra genin dizisini bir RNA zinciri olarak kopyalar, ta ki terminatör (sonlayıcı, İng. 'terminator') adlı bir DNA bölgesine gelip orada durup DNA'dan kopana kadar. DNA bağımlı DNA polimeraz da olduğu gibi, RNA polimeraz II (ökaryotlardaki çoğu genin transkripsiyonun yapan enzim) de çeşitli düzenleyici ve yardımcı proteinlerden oluşmuş büyük bir protein kompleksinin parçası olarak çalışır.[85]
Genetik Rekombinasyon
 |
 |
- Daha fazla bilgi: Genetik rekombinasyon
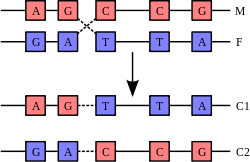
Bir DNA sarmalı genelde başka DNA parçaları ile etkileşmez, ve hatta insan hücrelerinde farklı kromozomlar çekirdekte farklı bölgelerde yer alırlar.[87] Farklı kromozomların fiziksel olarak bu şekilde ayrı tutulması DNA'nın kararlı bir bilgi deposu olarak işlev görmesinde önemli bir rol oynar. Kromozomların birbiriyle etkileştiği zamanlar sadece rekombinasyona girdikleri krosover sırasındadır. Krosover sırasında iki DNA sarmalı kesilir, bir bölüm yer değiştirir ve kesik uçlar birleşir.
Rekombinasyon sayesinde kromozomlar arasında genetik bilgi takası olur ve yeni gen kombinasyonları meydan gelir, bunun doğal seleksiyonun verimini artırdığı ve yeni proteinlerin hızlı evrimleşmesinde önemli olduğu düşünülmektedir.[88] Genetik rekombinasyon DNA tamiriyle de ilişkilidir, özellikle çift iplikli kırılmalara hücrenin tepkisinde.[89]
Kromozom sarılmasının en yaygın şekli homolog rekombinasyondur, bunda iki kromozom birbirine çok benzer dizilere sahiptir. Non-homolog rekombinasyon hücreye zarar verici olabilir çünkü kromozomal translokasyon ve genetik anormalliklere yol açabilir. Rekombinasyon tepkimesi rekombinaz olarak adlandırılan enzimler (örneğin RAD51) tarafından katalizlenir.[90] Rekombinasyonun ilk adımı çift iplikli bir kesik oluşturulmasıdır, bu ya bir endonükleaz ya da DNA hasarı sonucunda meydana gelir.[91] Rekombinaz tarafından kısmen katalizlenen bir dizi adım sonucunda iki sarmal en az bir Holliday bağlantısı tarafından birleştirilir: her sarmalın bir ipliği, öbür sarmalda ona komplementer olan öbür iplik ile kaynaşır. Holliday bağlantısı, tetrahedral bir yapıdır, bu şekilde birleşmiş iki kromozomda bir ipliğin bir diğeriyle yer değiştirmesiyle bu yapı kromozomlar boyunca ilerler. Rekombinasyon tepkimesi, bağlantının kesilmesi ve serbest kalan DNA uçlarının tekrar birleşmesi ile son bulur.[92]
DNA metabolizmasının evrimi
- Daha fazla bilgi: RNA dünya hipotezi
DNA'da bulunan genetik bilgi tüm modern canlıların işlev görmesine, yani büyümesi ve çoğalmasına olanak sağlar. Ancak, 4 milyar yıldır sürmekte olan yaşamın tarihçesi boyunca DNA'nın bu işlevi yerine getirdiği belli değildir, yaşamın en eski biçimlerinin kullanmış olduğu kalıtsal malzemenin RNA olduğu öne sürülmüştür.[81][93] RNA, hem genetik bilgi aktarma hem de ribozimlerin parçası olarak katalizör özelliğine sahip olmasından dolayı ilk hücrelerin metabolizmasında merkezî bir rol oynamış olabilir.[94] Nükleik asitlerin hem kalıtımda hem de katalizde rol oynadığı bu eski RNA dünyası, günümüz genetik kodunun dört nükleotit bazından oluşmuş şekilde evrimleşmesine etki etmiş olabilir. Bunun nedeni, bir canlıdaki bazların sayısının azlığının replikasyon verimini artıracağı ama bazların çokluğunun ise ribozimlerin katalitik verimini artıracağı, bu iki zıt etki ile kalıtsal bilgiyi kodlayan baz sayısının dört olarak dengelenmiş olabileceği öne sürülmüştür.[95]
Ne var ki, eski genetik sistemler hakkında doğrudan delil mevcut değildir, çünkü çoğu fosillerden DNA elde edilmesi mümkün değildir. Bunun nedeni, çevre etkilerine maruz kalan DNA'nın bir milyon yıldan az süre dayanması ve çözelti içinde zamanla küçük parçalara yıkımıdır.[96] Eski DNA'nın izole edilmiş olduğuna dair iddialar vardır, özellikle 250 milyon evvelden kalma bir tuz kristalı içinde canlı kalmış bir bakterinin izole edildiği iddia edilmiştir[97] ama bu iddialar tartışmalıdır.[98][99]
Teknolojide kullanım
Gen mühendisliği
- Daha fazla bilgi: Moleküler biyoloji ve gen mühendisliği
Modern biyoloji ve biyokimyada rekombinant DNA teknolojisi yoğun bir şekilde kullanılır. Rekombinant DNA başka DNA parçalarından bir araya getirilmiş yapay bir DNA'dır. DNA parçaları, plazmit veya viral vektörler aracılığıyla canlıların içine transformasyon yoluyla sokulabilir.[100] Bu yolla ortaya çıkan, genetik değişime uğramış canlılar kullanılarak rekombinant proteinler üretilebilir, bunlar tibbi araştırmalarda[101] veya tarımda[102][103] kullanılabilir.
Adlî bilim
- Daha fazla bilgi: DNA profillemesi
Adli bilimciler, bir suç mahalinde bulunmuş kan, meni, deri, tükürük veya saçta bulunan DNA'yı kullanarak bir failin kimliğini belirleyebilirler. Bu işleme genetik parmak izi çıkarma veya genetik profilleme denir. DNA profillemesinde, tekrarlı diziler (mikrosatelit ve minisatelit) içeren DNA'nın değişken kısımlarının uzunlukları belirlenir, bunlar farklı insanlarda karşılaştırılır. Bu yöntem bir suçlunun tanınması için son derece güvenilir bir yöntemdir.[104] Ancak, eğer suç mahaline birde fazla kişinin DNA'sı bulaşmışsa bu kimlik belirleme karmaşıklaşabilir.[105] DNA profillemesi 1984'te Britanyalı genetikçi Sir Alec Jeffreys,[106] tarafından geliştirilmiş ve adli bilimde ilk defa 1988'de Enderby cinayetleri için Colin Pitchfork'un suçlu bulnmasında kullanılmıştır.[107] Bazı tür suçları işlemiş kişiler bir veritabanında depolanmak amacıyla kendi DNA'larından bir örnek vermeye mecbur tutulabilirler. Bu sayede suç mahalinde bulunmuş DNA örneğinden başka elde hiçbir delil bulunmayan bazı eski vakalar çözülebilmiştir. DNA profillemesi katliam kurbanlarının kimliklerinin belirlenmesinde de kullanılmıştır.[108]
Biyoenformatik
- Daha fazla bilgi: Biyoenformatik
DNA dizilerinin bilgisayar aracılığıyla işlenmesi, aranması ve analizi, biyoenformatik bilminin konuları arasındadır. DNA dizilerinin depolanması ve aranması için yöntemlerin geliştirilmesi sayesinde bilgisayar bilimlerinde önemli ilerlemeler katedilmiştir, özelikle dizi arama algoritmaları, makine öğrenimi ve veritabanı teorisi konularında.[109] Dizi arama ve eşlendirme algoritmaları harflerden oluşan uzun diziler içinde daha kısa harf dizilerinin bulunmasıyla ilgilidir, bunlar belli nükleotit dizilerinin bulunması için geliştirilmiştir.[110] Yazı editörü programlarının kullandığı algoritmalar DNA dizileri durumunda son derece verimsiz çalışırlar, DNA dizilerini oluşturan farklı karakterlerin küçük sayısından dolayı. Bununla ilişkili olan dizi hizalama problemi ise benzer dizileri bulmayı ve bunları birbirinden faklı kılan mutasyonları tanımlamayı amaçlar. Bu teknikler, özellikle çoklu dizi hizalaması, filogenetik ilişki ve protein işlevi araştırmalarında kullanılır.[111] Bir genomun tamamına karşılık gelen DNA dizilerinin kullanılması için bu dizilerin üzerinde genlerin ve onların düzenleyici elemanlarının yerlerinin kaydedilmesi (İng. annotation) gerekmektedir. DNA dizilerinde protein veya RNA kodlayıcı genlerin özelliklerine sahip bölgelerin tanınması, gen bulma algoritmaları sayesinde mümkündür, bunlar sayesinde bilim insanları bir genin ürününü önceden tahmin edebilirler, bu ürün laboratuvarda daha saflaştırılmadan.[112]
DNA nanoteknolojisi
- Daha fazla bilgi: DNA nanoteknolojisi
DNA nanoteknolojisi DNA'ya has moleküler tanıma özelliklerini kullanarak faydalı özelliklere sahip, kendi kendini oluşturan, dallı DNA komplksleri imal eder. DNA böylede biyolojik bilgi taşımak için değil, yapısal bir malzeme olarak kullanılır. Bu yolla iki boyutlu periyodik dizilimler ve polihedral şekilli üç boyutlu yapılar yaratılmıştır. Nanomekanik araçlar ve algoritmik olarak oluşan yapılar da gösterilmiş, bu DNA yapıları ile başka moleküllerin (altın nano tanecikleri ve streptavidin proteinlerinin) düzenlenmesi sağlanabilmiştir.
Tarih ve antropoloji
- Daha fazla bilgi: Filogenetik ve Genetik soybilim
Zaman içinde DNA'da biriken mutasyonlar sonra kalıtsal olarak aktarıldığı için, taşıdığı bilgi bir anlamda tarihseldir. Genetikçiler DNA dizlerini karşılaştırarak bir canlının evrimsel tarihi yani onun filogenetiği hakında çıkarımlar yapabilirler.[113] Filogenetik sahası evimsel biyolojide güçlü bir araçtır. Bir türün bireylerine ait DNA dizileri karşılaştırıldığında topluluk genetikçileri o topluluğun tarihine dair bilgiler edinebilirler. Ekolojik genetikten antropolojiye kadar uzanan çeşitli sahalarda bu bilgilerden yararlanılabilir. Örneğin, Tevrat'ta söz konusu olan İsrail'in on kayıp kavmi, DNA bulguları ile tanımlanmaktadır.[114][115]
DNA ayrıca aile ilişkilerini belirlemek için kullanılmıştır, örneğin Amerikan başkanlarından Thomas Jefferson'un kölesi Sally Hemings'in soyundan kişiler ile Jefferson arasında akrabalık olduğunun kanıtlanmasında. Bu amaçlı kullanım, yukarda değinilen suç tahkikatlarında DNA'nın kullanılmasına benzerdir. Nitekim, bazı tahkikatların çözümlenmesi, suç mahalinde bulunan DNA'nın suçlunun akrabalarının DNA'sıyla uyuşması sayesinde olmuştur.[116]
DNA araştırmasının tarihçesi

- Daha fazla bilgi: Moleküler biyolojinin tarihçesi
DNA ilk İsviçreli hekim Friedrich Miescher tarafından saflaştırılmıştır, kendisi 1869'da atık cerrahi pansumanlardaki irin içinde mikroskopik bir madde keşfetmiştir. Hücre çekirdeklerinde (nükleus) bulunduğu için ona "nüklein" adını vermiştir.[117] 1919'da Phoebus Levene, nükleotit birimleri oluşturan baz, şeker ve fosfatı tanımlanmıştır.[118] Levene DNA'nın, birbirine fosfat grupları ile bağlı olan nükleotit birimlerden oluşan bir zincir olduğunu öne sürmüştür. Ancak, Levene, bu zincirin kısa olduğunu ve bazları kendini tekrar eden bir sıralamaya sahip olduğunu düşünmüştür. 1937'de William Astbury DNA'nın düzenli bir yapıya sahip olduğunu gösteren ilk X ışını difraksiyon görüntülerini elde etti.[119]
1928'de Frederick Griffith, Pnömokok bakterisinin "düz" şeklini belirleyen özelliğin "buruşuk" şekilli Pnömokok bakterilere aktarılmasının mümkün olduğunu, bunun için ölü "düz" bakterilerin canlı "buruşuk" bakterilerle karıştırılmasının yettiğini gösterdi.[120] Bu deneysel sistem kullanarak Oswald Avery ve arkadaşları Colin MacLeod ve Maclyn McCarty 1943'de değiştirici etmenin DNA olduğunu gösterdiler.[121] 1952'de Alfred Hershey ve Martha Chase tarafından Hershey-Chase deneyinde T2 fajının genetik malzemesinin DNA olduğunu göstererek DNA kalıtımdaki rolü teyid ettiler.[122]

1953'te James D. Watson ve Francis Crick DNA'nın bugün kabul görmüş yapısını Nature dergisinde öne sürdüler.[123] Çift sarmallı moleküler modelleri tek bir X-ışını kırınım resmine dayanmaktaydı, bu resim Rosalind Franklin ve Raymond Gosling tarafından Mayıs 1952'de elde edilmişti. Modellerini dayandırdıkları bir diğer bilgi Erwin Chargaff'ın evvelki yıllarda kendilerine özel olarak iletmiş olduğu, DNA bazlarını birbiriyle eşleştiğiydi. Chargaff kuralları hem B-DNA'nın hem de A-DNA'nın çifte sarmallı biçimini tespit etmekte önemli bir rol oynamıştır.
Watson ve Crick modelinin destekleyen deneysel kanıtlar Nature dergisinin aynı sayısında yayımlanan beş makalede yer aldı.[124] Bunlardan Franklin ve Gosling'in makalesi, Watson ve Crick modelini kısmen destekleyen, kendi X-ışını kırınım verileri ve analiz yönteminin ilk yayımlanmasıydı.[125][126] Dergini aynı sayısında DNA yapısı hakkında Maurice Wilkins ve iki arkadaşının bir makalesi vardı, onların in vivo B-DNA X-ışını kırınım örüntüleri üzerinde yaptıkları analizler, iki sayfa geride Crick ve Watson tarafından önerilen çifte sarmal modelini destekliyordu.[127] 1962'de Franklin'in ölümünden sonra Watson, Crick ve Wilkins birlikte Nobel Fizyoloji veya Tıp Ödülü'nü kazandılar.[128] O zamanki Nobel ödülleri ancak hayatta olan kişilere ödülün vermesine izin veriyordu. Keşif için kimlerin kredi alması gerektiği hakkında tartışma devam etmektedir.[129]
Crick, 1957'de yaptığı etkili bir sunumda, moleküler biyolojinin "Temel Dogması"nı ortaya koyarak DNA, RNA ve proteinler arasındaki ilişkiyi, bu konuda kanıtlar henüz tamamen toplanmadan, özetledi, ayrıca "adaptör hipotezi"ni dile getirdi.[130] Çift sarmallı yapının ima ettiği kopyalama mekanizmasının teyidi, 1958'de yayımlanan Meselson-Stahl deneyi ile edildi.[131] Crick ve arkadaşları tarafından yapılan diğer çalışmalar genetik kodun, kodon olarak adlandırılan, örtüşmeyen baz üçlülerinden oluştuğunu gösterdi, bu sayede Har Gobind Khorana, Robert W. Holley ve Marshall Warren Nirenberg genetik kodu çözdüler.[132] Bu keşifler moleküler biyolojinin doğumuna karşılık gelir.
Ayrıca bakınız
- Kristalografi
- DNA mikroçip
- DNA dizileme
- Üç sarmallı DNA
- Genetik mühendisliği
- A-DNA
- X-ışını kırınımı
- Southern blot
Notlar
- 1 2 Alberts, Bruce; Alexander Johnson, Julian Lewis, Martin Raff, Keith Roberts, and Peter Walters (2002). Molecular Biology of the Cell; Fourth Edition. New York and London: Garland Science. ISBN 0-8153-3218-1. http://www.ncbi.nlm.nih.gov/books/bv.fcgi?call=bv.View..ShowTOC&rid=mboc4.TOC&depth=2.
- ↑ Butler, John M. (2001). Forensic DNA Typing. Elsevier. ISBN 978-0-12-147951-0.pp. 14–15.
- ↑ Mandelkern M, Elias J, Eden D, Crothers D (1981). "The dimensions of DNA in solution". J Mol Biol 152 (1): 153–61. DOI:10.1016/0022-2836(81)90099-1. PMID 7338906.
- ↑ Gregory S, et al. (2006). "The DNA sequence and biological annotation of human chromosome 1". Nature 441 (7091): 315–21. DOI:10.1038/nature04727. PMID 16710414.
- ↑ Watson J, Crick F (1953). "Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid". Nature 171 (4356): 737–8. DOI:10.1038/171737a0. PMID 13054692. http://profiles.nlm.nih.gov/SC/B/B/Y/W/_/scbbyw.pdf.
- 1 2 Berg J., Tymoczko J. and Stryer L. (2002) Biochemistry. W. H. Freeman and Company ISBN 0-7167-4955-6
- ↑ Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents IUPAC-IUB Commission on Biochemical Nomenclature (CBN), Accessed 03 Jan 2006
- 1 2 Ghosh A, Bansal M (2003). "A glossary of DNA structures from A to Z". Acta Crystallogr D Biol Crystallogr 59 (Pt 4): 620–6. DOI:10.1107/S0907444903003251. PMID 12657780.
- ↑ Wing R, Drew H, Takano T, Broka C, Tanaka S., Itakura K, Dickerson R (1980). "Crystal structure analysis of a complete turn of B-DNA". Nature 287 (5784): 755–8. DOI:10.1038/287755a0. PMID 7432492.
- ↑ Pabo C, Sauer R (1984). "Protein-DNA recognition". Annu Rev Biochem 53: 293–321. DOI:10.1146/annurev.bi.53.070184.001453. PMID 6236744.
- ↑ Ponnuswamy P, Gromiha M (1994). "On the conformational stability of oligonucleotide duplexes and tRNA molecules". J Theor Biol 169 (4): 419–32. DOI:10.1006/jtbi.1994.1163. PMID 7526075.
- ↑ Clausen-Schaumann H, Rief M, Tolksdorf C, Gaub H (2000). "Mechanical stability of single DNA molecules". Biophys J 78 (4): 1997–2007. PMID 10733978. http://www.pubmedcentral.nih.gov/picrender.fcgi?artid=1300792&blobtype=pdf.
- ↑ Chalikian T, Völker J, Plum G, Breslauer K (1999). "A more unified picture for the thermodynamics of nucleic acid duplex melting: a characterization by calorimetric and volumetric techniques". Proc Natl Acad Sci USA 96 (14): 7853–8. DOI:10.1073/pnas.96.14.7853. PMID 10393911. http://www.pubmedcentral.nih.gov/picrender.fcgi?artid=22151&blobtype=pdf.
- ↑ deHaseth P, Helmann J (1995). "Open complex formation by Escherichia coli RNA polymerase: the mechanism of polymerase-induced strand separation of double helical DNA". Mol Microbiol 16 (5): 817–24. DOI:10.1111/j.1365-2958.1995.tb02309.x. PMID 7476180.
- ↑ Isaksson J, Acharya S, Barman J, Cheruku P, Chattopadhyaya J (2004). "Single-stranded adenine-rich DNA and RNA retain structural characteristics of their respective double-stranded conformations and show directional differences in stacking pattern". Biochemistry 43 (51): 15996–6010. DOI:10.1021/bi048221v. PMID 15609994.
- ↑ Designation of the two strands of DNA JCBN/NC-IUB Newsletter 1989, Accessed 07 May 2008
- ↑ Hüttenhofer A, Schattner P, Polacek N (2005). "Non-coding RNAs: hope or hype?". Trends Genet 21 (5): 289–97. DOI:10.1016/j.tig.2005.03.007. PMID 15851066.
- ↑ Munroe S (2004). "Diversity of antisense regulation in eukaryotes: multiple mechanisms, emerging patterns". J Cell Biochem 93 (4): 664–71. DOI:10.1002/jcb.20252. PMID 15389973.
- ↑ Makalowska I, Lin C, Makalowski W (2005). "Overlapping genes in vertebrate genomes". Comput Biol Chem 29 (1): 1–12. DOI:10.1016/j.compbiolchem.2004.12.006. PMID 15680581.
- ↑ Johnson Z, Chisholm S (2004). "Properties of overlapping genes are conserved across microbial genomes". Genome Res 14 (11): 2268–72. DOI:10.1101/gr.2433104. PMID 15520290.
- ↑ Lamb R, Horvath C (1991). "Diversity of coding strategies in influenza viruses". Trends Genet 7 (8): 261–6. PMID 1771674.
- ↑ Benham C, Mielke S (2005). "DNA mechanics". Annu Rev Biomed Eng 7: 21–53. DOI:10.1146/annurev.bioeng.6.062403.132016. PMID 16004565.
- 1 2 Champoux J (2001). "DNA topoisomerases: structure, function, and mechanism". Annu Rev Biochem 70: 369–413. DOI:10.1146/annurev.biochem.70.1.369. PMID 11395412.
- 1 2 Wang J (2002). "Cellular roles of DNA topoisomerases: a molecular perspective". Nat Rev Mol Cell Biol 3 (6): 430–40. DOI:10.1038/nrm831. PMID 12042765.
- ↑ Basu H, Feuerstein B, Zarling D, Shafer R, Marton L (1988). "Recognition of Z-RNA and Z-DNA determinants by polyamines in solution: experimental and theoretical studies". J Biomol Struct Dyn 6 (2): 299–309. PMID 2482766.
- ↑ Leslie AG, Arnott S, Chandrasekaran R, Ratliff RL (1980). "Polymorphism of DNA double helices". J. Mol. Biol. 143 (1): 49–72. DOI:10.1016/0022-2836(80)90124-2. PMID 7441761.
- ↑ Wahl M, Sundaralingam M (1997). "Crystal structures of A-DNA duplexes". Biopolymers 44 (1): 45–63. DOI:10.1002/(SICI)1097-0282(1997)44:1. PMID 9097733.
- ↑ Lu XJ, Shakked Z, Olson WK (2000). "A-form conformational motifs in ligand-bound DNA structures". J. Mol. Biol. 300 (4): 819–40. DOI:10.1006/jmbi.2000.3690. PMID 10891271.
- ↑ Rothenburg S, Koch-Nolte F, Haag F (2001). "DNA methylation and Z-DNA formation as mediators of quantitative differences in the expression of alleles". Immunol Rev 184: 286–98. DOI:10.1034/j.1600-065x.2001.1840125.x. PMID 12086319.
- ↑ Oh D, Kim Y, Rich A (2002). "Z-DNA-binding proteins can act as potent effectors of gene expression in vivo". Proc. Natl. Acad. Sci. U.S.A. 99 (26): 16666–71. DOI:10.1073/pnas.262672699. PMID 12486233. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pubmed&pubmedid=12486233.
- ↑ Created from NDB UD0017 koordinatlarından üretilmiştir
- 1 2 Greider C, Blackburn E (1985). "Identification of a specific telomere terminal transferase activity in Tetrahymena extracts". Cell 43 (2 Pt 1): 405–13. DOI:10.1016/0092-8674(85)90170-9. PMID 3907856.
- 1 2 3 Nugent C, Lundblad V (1998). "The telomerase reverse transcriptase: components and regulation". Genes Dev 12 (8): 1073–85. DOI:10.1101/gad.12.8.1073. PMID 9553037. http://www.genesdev.org/cgi/content/full/12/8/1073.
- ↑ Wright W, Tesmer V, Huffman K, Levene S, Shay J (1997). "Normal human chromosomes have long G-rich telomeric overhangs at one end". Genes Dev 11 (21): 2801–9. DOI:10.1101/gad.11.21.2801. PMID 9353250. http://www.genesdev.org/cgi/content/full/11/21/2801.
- 1 2 Burge S, Parkinson G, Hazel P, Todd A, Neidle S (2006). "Quadruplex DNA: sequence, topology and structure". Nucleic Acids Res 34 (19): 5402–15. DOI:10.1093/nar/gkl655. PMID 17012276. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pubmed&pubmedid=17012276.
- ↑ Parkinson G, Lee M, Neidle S (2002). "Crystal structure of parallel quadruplexes from human telomeric DNA". Nature 417 (6891): 876–80. DOI:10.1038/nature755. PMID 12050675.
- ↑ Griffith J, Comeau L, Rosenfield S, Stansel R, Bianchi A, Moss H, de Lange T (1999). "Mammalian telomeres end in a large duplex loop". Cell 97 (4): 503–14. DOI:10.1016/S0092-8674(00)80760-6. PMID 10338214.
- ↑ Klose R, Bird A (2006). "Genomic DNA methylation: the mark and its mediators". Trends Biochem Sci 31 (2): 89–97. DOI:10.1016/j.tibs.2005.12.008. PMID 16403636.
- ↑ Bird A (2002). "DNA methylation patterns and epigenetic memory". Genes Dev 16 (1): 6–21. DOI:10.1101/gad.947102. PMID 11782440.
- ↑ Walsh C, Xu G (2006). "Cytosine methylation and DNA repair". Curr Top Microbiol Immunol 301: 283–315. DOI:10.1007/3-540-31390-7_11. PMID 16570853.
- ↑ Ratel D, Ravanat J, Berger F, Wion D (2006). "N6-methyladenine: the other methylated base of DNA". Bioessays 28 (3): 309–15. DOI:10.1002/bies.20342. PMID 16479578.
- ↑ Gommers-Ampt J, Van Leeuwen F, de Beer A, Vliegenthart J, Dizdaroglu M, Kowalak J, Crain P, Borst P (1993). "beta-D-glucosyl-hydroxymethyluracil: a novel modified base present in the DNA of the parasitic protozoan T. brucei". Cell 75 (6): 1129–36. DOI:10.1016/0092-8674(93)90322-H. PMID 8261512.
- ↑ PDB 1JDG koordinatlarından üretilmiştir
- ↑ Douki T, Reynaud-Angelin A, Cadet J, Sage E (2003). "Bipyrimidine photoproducts rather than oxidative lesions are the main type of DNA damage involved in the genotoxic effect of solar UVA radiation". Biochemistry 42 (30): 9221–6. DOI:10.1021/bi034593c. PMID 12885257.
- ↑ Cadet J, Delatour T, Douki T, Gasparutto D, Pouget J, Ravanat J, Sauvaigo S (1999). "Hydroxyl radicals and DNA base damage". Mutat Res 424 (1–2): 9–21. PMID 10064846.
- ↑ Shigenaga M, Gimeno C, Ames B (1989). "Urinary 8-hydroxy-2′-deoxyguanosine as a biological marker of in vivo oxidative DNA damage". Proc Natl Acad Sci USA 86 (24): 9697–701. DOI:10.1073/pnas.86.24.9697. PMID 2602371. http://www.pnas.org/cgi/reprint/86/24/9697.
- ↑ Cathcart R, Schwiers E, Saul R, Ames B (1984). "Thymine glycol and thymidine glycol in human and rat urine: a possible assay for oxidative DNA damage". Proc Natl Acad Sci USA 81 (18): 5633–7. DOI:10.1073/pnas.81.18.5633. PMID 6592579. http://www.pnas.org/cgi/reprint/81/18/5633.pdf.
- ↑ Valerie K, Povirk L (2003). "Regulation and mechanisms of mammalian double-strand break repair". Oncogene 22 (37): 5792–812. DOI:10.1038/sj.onc.1206679. PMID 12947387.
- ↑ Ferguson L, Denny W (1991). "The genetic toxicology of acridines". Mutat Res 258 (2): 123–60. PMID 1881402.
- ↑ Jeffrey A (1985). "DNA modification by chemical carcinogens". Pharmacol Ther 28 (2): 237–72. DOI:10.1016/0163-7258(85)90013-0. PMID 3936066.
- ↑ Stephens T, Bunde C, Fillmore B (2000). "Mechanism of action in thalidomide teratogenesis". Biochem Pharmacol 59 (12): 1489–99. DOI:10.1016/S0006-2952(99)00388-3. PMID 10799645.
- ↑ Braña M, Cacho M, Gradillas A, de Pascual-Teresa B, Ramos A (2001). "Intercalators as anticancer drugs". Curr Pharm Des 7 (17): 1745–80. DOI:10.2174/1381612013397113. PMID 11562309.
- ↑ Venter J, et al. (2001). "The sequence of the human genome". Science 291 (5507): 1304–51. DOI:10.1126/science.1058040. PMID 11181995.
- ↑ Thanbichler M, Wang S, Shapiro L (2005). "The bacterial nucleoid: a highly organized and dynamic structure". J Cell Biochem 96 (3): 506–21. DOI:10.1002/jcb.20519. PMID 15988757.
- ↑ Wolfsberg T, McEntyre J, Schuler G (2001). "Guide to the draft human genome". Nature 409 (6822): 824–6. DOI:10.1038/35057000. PMID 11236998.
- ↑ Gregory T (2005). "The C-value enigma in plants and animals: a review of parallels and an appeal for partnership". Ann Bot (Lond) 95 (1): 133–46. DOI:10.1093/aob/mci009. PMID 15596463. http://aob.oxfordjournals.org/cgi/content/full/95/1/133.
- ↑ The ENCODE Project Consortium (2007). "Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project". Nature 447 (7146): 799–816. DOI:10.1038/nature05874.
- ↑ PDB 1MSW koordinatlarından üretilmiştir
- ↑ Pidoux A, Allshire R (2005). "The role of heterochromatin in centromere function". Philos Trans R Soc Lond B Biol Sci 360 (1455): 569–79. DOI:10.1098/rstb.2004.1611. PMID 15905142. http://www.pubmedcentral.nih.gov/articlerender.fcgi?artid=1569473.
- ↑ Harrison P, Hegyi H, Balasubramanian S, Luscombe N, Bertone P, Echols N, Johnson T, Gerstein M (2002). "Molecular fossils in the human genome: identification and analysis of the pseudogenes in chromosomes 21 and 22". Genome Res 12 (2): 272–80. DOI:10.1101/gr.207102. PMID 11827946. http://www.genome.org/cgi/content/full/12/2/272.
- ↑ Harrison P, Gerstein M (2002). "Studying genomes through the aeons: protein families, pseudogenes and proteome evolution". J Mol Biol 318 (5): 1155–74. DOI:10.1016/S0022-2836(02)00109-2. PMID 12083509.
- ↑ Albà M (2001). "Replicative DNA polymerases". Genome Biol 2 (1): REVIEWS3002. DOI:10.1186/gb-2001-2-1-reviews3002. PMID 11178285. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pubmed&pubmedid=11178285.
- ↑ Sandman K, Pereira S, Reeve J (1998). "Diversity of prokaryotic chromosomal proteins and the origin of the nucleosome". Cell Mol Life Sci 54 (12): 1350–64. DOI:10.1007/s000180050259. PMID 9893710.
- ↑ Dame RT (2005). "The role of nucleoid-associated proteins in the organization and compaction of bacterial chromatin". Mol. Microbiol. 56 (4): 858–70. DOI:10.1111/j.1365-2958.2005.04598.x. PMID 15853876.
- ↑ Luger K, Mäder A, Richmond R, Sargent D, Richmond T (1997). "Crystal structure of the nucleosome core particle at 2.8 A resolution". Nature 389 (6648): 251–60. DOI:10.1038/38444. PMID 9305837.
- ↑ Jenuwein T, Allis C (2001). "Translating the histone code". Science 293 (5532): 1074–80. DOI:10.1126/science.1063127. PMID 11498575.
- ↑ Ito T. "Nucleosome assembly and remodelling". Curr Top Microbiol Immunol 274: 1–22. PMID 12596902.
- ↑ Thomas J (2001). "HMG1 and 2: architectural DNA-binding proteins". Biochem Soc Trans 29 (Pt 4): 395–401. DOI:10.1042/BST0290395. PMID 11497996.
- ↑ Grosschedl R, Giese K, Pagel J (1994). "HMG domain proteins: architectural elements in the assembly of nucleoprotein structures". Trends Genet 10 (3): 94–100. DOI:10.1016/0168-9525(94)90232-1. PMID 8178371.
- ↑ Iftode C, Daniely Y, Borowiec J (1999). "Replication protein A (RPA): the eukaryotic SSB". Crit Rev Biochem Mol Biol 34 (3): 141–80. DOI:10.1080/10409239991209255. PMID 10473346.
- ↑ PDB 1LMB koordinatlarından üretilmiştir
- ↑ Myers L, Kornberg R (2000). "Mediator of transcriptional regulation". Annu Rev Biochem 69: 729–49. DOI:10.1146/annurev.biochem.69.1.729. PMID 10966474.
- ↑ Spiegelman B, Heinrich R (2004). "Biological control through regulated transcriptional coactivators". Cell 119 (2): 157–67. DOI:10.1016/j.cell.2004.09.037. PMID 15479634.
- ↑ Li Z, Van Calcar S, Qu C, Cavenee W, Zhang M, Ren B (2003). "A global transcriptional regulatory role for c-Myc in Burkitt's lymphoma cells". Proc Natl Acad Sci USA 100 (14): 8164–9. DOI:10.1073/pnas.1332764100. PMID 12808131. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pubmed&pubmedid=12808131.
- ↑ Pabo C, Sauer R (1984). "Protein-DNA recognition". Annu Rev Biochem 53: 293–321. DOI:10.1146/annurev.bi.53.070184.001453. PMID 6236744.
- ↑ PDB 1RVA koordinatlarından yaratılmıştır
- ↑ Bickle T, Krüger D (1993). "Biology of DNA restriction". Microbiol Rev 57 (2): 434–50. PMID 8336674. http://www.pubmedcentral.nih.gov/picrender.fcgi?artid=372918&blobtype=pdf.
- ↑ Doherty A, Suh S (2000). "Structural and mechanistic conservation in DNA ligases". Nucleic Acids Res 28 (21): 4051–8. DOI:10.1093/nar/28.21.4051. PMID 11058099. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pubmed&pubmedid=11058099.
- ↑ Schoeffler A, Berger J (2005). "Recent advances in understanding structure-function relationships in the type II topoisomerase mechanism". Biochem Soc Trans 33 (Pt 6): 1465–70. DOI:10.1042/BST20051465. PMID 16246147.
- ↑ Tuteja N, Tuteja R (2004). "Unraveling DNA helicases. Motif, structure, mechanism and function". Eur J Biochem 271 (10): 1849–63. DOI:10.1111/j.1432-1033.2004.04094.x. PMID 15128295.
- 1 2 Joyce C, Steitz T (1995). "Polymerase structures and function: variations on a theme?". J Bacteriol 177 (22): 6321–9. PMID 7592405. http://www.pubmedcentral.nih.gov/picrender.fcgi?artid=177480&blobtype=pdf.
- ↑ Hubscher U, Maga G, Spadari S (2002). "Eukaryotic DNA polymerases". Annu Rev Biochem 71: 133–63. DOI:10.1146/annurev.biochem.71.090501.150041. PMID 12045093.
- ↑ Johnson A, O'Donnell M (2005). "Cellular DNA replicases: components and dynamics at the replication fork". Annu Rev Biochem 74: 283–315. DOI:10.1146/annurev.biochem.73.011303.073859. PMID 15952889.
- ↑ Tarrago-Litvak L, Andréola M, Nevinsky G, Sarih-Cottin L, Litvak S (1994). "The reverse transcriptase of HIV-1: from enzymology to therapeutic intervention". FASEB J 8 (8): 497–503. PMID 7514143. http://www.fasebj.org/cgi/reprint/8/8/497.
- ↑ Martinez E (2002). "Multi-protein complexes in eukaryotic gene transcription". Plant Mol Biol 50 (6): 925–47. DOI:10.1023/A:1021258713850. PMID 12516863.
- ↑ PDB 1M6G kordinatlarından üretilmiştir
- ↑ Cremer T, Cremer C (2001). "Chromosome territories, nuclear architecture and gene regulation in mammalian cells". Nat Rev Genet 2 (4): 292–301. DOI:10.1038/35066075. PMID 11283701.
- ↑ Pál C, Papp B, Lercher M (2006). "An integrated view of protein evolution". Nat Rev Genet 7 (5): 337–48. DOI:10.1038/nrg1838. PMID 16619049.
- ↑ O'Driscoll M, Jeggo P (2006). "The role of double-strand break repair - insights from human genetics". Nat Rev Genet 7 (1): 45–54. DOI:10.1038/nrg1746. PMID 16369571.
- ↑ Vispé S, Defais M (1997). "Mammalian Rad51 protein: a RecA homologue with pleiotropic functions". Biochimie 79 (9-10): 587–92. DOI:10.1016/S0300-9084(97)82007-X. PMID 9466696.
- ↑ Neale MJ, Keeney S (2006). "Clarifying the mechanics of DNA strand exchange in meiotic recombination". Nature 442 (7099): 153–8. DOI:10.1038/nature04885. PMID 16838012.
- ↑ Dickman M, Ingleston S, Sedelnikova S, Rafferty J, Lloyd R, Grasby J, Hornby D (2002). "The RuvABC resolvasome". Eur J Biochem 269 (22): 5492–501. DOI:10.1046/j.1432-1033.2002.03250.x. PMID 12423347.
- ↑ Orgel L. "Prebiotic chemistry and the origin of the RNA world". Crit Rev Biochem Mol Biol 39 (2): 99–123. DOI:10.1080/10409230490460765. PMID 15217990. http://www.crbmb.com/cgi/reprint/39/2/99.pdf.
- ↑ Davenport R (2001). "Ribozymes. Making copies in the RNA world". Science 292 (5520): 1278. DOI:10.1126/science.292.5520.1278a. PMID 11360970.
- ↑ Szathmáry E (1992). "What is the optimum size for the genetic alphabet?". Proc Natl Acad Sci USA 89 (7): 2614–8. DOI:10.1073/pnas.89.7.2614. PMID 1372984. http://www.pnas.org/cgi/reprint/89/7/2614.pdf.
- ↑ Lindahl T (1993). "Instability and decay of the primary structure of DNA". Nature 362 (6422): 709–15. DOI:10.1038/362709a0. PMID 8469282.
- ↑ Vreeland R, Rosenzweig W, Powers D (2000). "Isolation of a 250 million-year-old halotolerant bacterium from a primary salt crystal". Nature 407 (6806): 897–900. DOI:10.1038/35038060. PMID 11057666.
- ↑ Hebsgaard M, Phillips M, Willerslev E (2005). "Geologically ancient DNA: fact or artefact?". Trends Microbiol 13 (5): 212–20. DOI:10.1016/j.tim.2005.03.010. PMID 15866038.
- ↑ Nickle D, Learn G, Rain M, Mullins J, Mittler J (2002). "Curiously modern DNA for a "250 million-year-old" bacterium". J Mol Evol 54 (1): 134–7. DOI:10.1007/s00239-001-0025-x. PMID 11734907.
- ↑ Goff SP, Berg P (1976). "Construction of hybrid viruses containing SV40 and lambda phage DNA segments and their propagation in cultured monkey cells". Cell 9 (4 PT 2): 695–705. DOI:10.1016/0092-8674(76)90133-1. PMID 189942.
- ↑ Houdebine L. "Transgenic animal models in biomedical research". Methods Mol Biol 360: 163–202. PMID 17172731.
- ↑ Daniell H, Dhingra A (2002). "Multigene engineering: dawn of an exciting new era in biotechnology". Curr Opin Biotechnol 13 (2): 136–41. DOI:10.1016/S0958-1669(02)00297-5. PMID 11950565.
- ↑ Job D (2002). "Plant biotechnology in agriculture". Biochimie 84 (11): 1105–10. DOI:10.1016/S0300-9084(02)00013-5. PMID 12595138.
- ↑ Collins A, Morton N (1994). "Likelihood ratios for DNA identification". Proc Natl Acad Sci USA 91 (13): 6007–11. DOI:10.1073/pnas.91.13.6007. PMID 8016106. http://www.pnas.org/cgi/reprint/91/13/6007.pdf.
- ↑ Weir B, Triggs C, Starling L, Stowell L, Walsh K, Buckleton J (1997). "Interpreting DNA mixtures". J Forensic Sci 42 (2): 213–22. PMID 9068179.
- ↑ Jeffreys A, Wilson V, Thein S (1985). "Individual-specific 'fingerprints' of human DNA". Nature 316 (6023): 76–9. DOI:10.1038/316076a0. PMID 2989708.
- ↑ Colin Pitchfork — first murder conviction on DNA evidence also clears the prime suspect Forensic Science Service Accessed 23 Dec 2006
- ↑ "DNA Identification in Mass Fatality Incidents". National Institute of Justice. September 2006. 24 Nisan 2014 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20140424165535/http://massfatality.dna.gov/Introduction/.
- ↑ Baldi, Pierre. Brunak, Soren (2001). Bioinformatics: The Machine Learning Approach. MIT Press. ISBN 978-0-262-02506-5.
- ↑ Gusfield, Dan. Algorithms on Strings, Trees, and Sequences: Computer Science and Computational Biology. Cambridge University Press, 15 January 1997. ISBN 978-0-521-58519-4.
- ↑ Sjölander K (2004). "Phylogenomic inference of protein molecular function: advances and challenges". Bioinformatics 20 (2): 170–9. DOI:10.1093/bioinformatics/bth021. PMID 14734307. http://bioinformatics.oxfordjournals.org/cgi/reprint/20/2/170.
- ↑ Mount DM (2004). Bioinformatics: Sequence and Genome Analysis (2 bas.). Cold Spring Harbor, NY: Cold Spring Harbor Laboratory Press. ISBN 0879697121.
- ↑ Wray G (2002). "Dating branches on the tree of life using DNA". Genome Biol 3 (1): REVIEWS0001. DOI:10.1046/j.1525-142X.1999.99010.x. PMID 11806830. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pubmed&pubmedid=11806830.
- ↑ Lost Tribes of Israel, NOVA, PBS airdate: 22 February 2000. Transcript available from PBS.org, (last accessed on 4 March 2006)
- ↑ Kleiman, Yaakov. "The Cohanim/DNA Connection: The fascinating story of how DNA studies confirm an ancient biblical tradition". aish.com (January 13, 2000). Accessed 4 March 2006.
- ↑ Bhattacharya, Shaoni. "Killer convicted thanks to relative's DNA". newscientist.com (20 April 2004). Erişim tarihi 22/12/2006
- ↑ Dahm R (2005). "Friedrich Miescher and the discovery of DNA". Dev Biol 278 (2): 274–88. DOI:10.1016/j.ydbio.2004.11.028. PMID 15680349.
- ↑ Levene P, (1919). "The structure of yeast nucleic acid". J Biol Chem 40 (2): 415–24. http://www.jbc.org/cgi/reprint/40/2/415.
- ↑ Astbury W, (1947). "Nucleic acid". Symp. SOC. Exp. Bbl 1 (66).
- ↑ Lorenz MG, Wackernagel W (1994). "Bacterial gene transfer by natural genetic transformation in the environment". Microbiol. Rev. 58 (3): 563–602. PMID 7968924. http://www.pubmedcentral.nih.gov/articlerender.fcgi?tool=pubmed&pubmedid=7968924.
- ↑ Avery O, MacLeod C, McCarty M (1944). "Studies on the chemical nature of the substance inducing transformation of pneumococcal types. Inductions of transformation by a desoxyribonucleic acid fraction isolated from pneumococcus type III". J Exp Med 79 (2): 137–158. DOI:10.1084/jem.79.2.137. http://www.jem.org/cgi/reprint/149/2/297.
- ↑ Hershey A, Chase M (1952). "Independent functions of viral protein and nucleic acid in growth of bacteriophage". J Gen Physiol 36 (1): 39–56. DOI:10.1085/jgp.36.1.39. PMID 12981234. http://www.jgp.org/cgi/reprint/36/1/39.pdf.
- ↑ Watson J.D. ve Crick F.H.C. (1953). "A Structure for Deoxyribose Nucleic Acid" (PDF). Nature 171 (4356): 737–738. DOI:10.1038/171737a0. PMID 13054692. http://www.nature.com/nature/dna50/watsoncrick.pdf.
- ↑ Nature Archives Double Helix of DNA: 50 Years
- ↑ Franklin, Rosalind ve Gosling, Raymond (1953). "Molecular Configuration in Sodium Thymonucleate. Franklin R. and Gosling R.G" (PDF). Nature 171 (4356): 740–1. DOI:10.1038/171740a0. PMID 13054694. http://www.nature.com/nature/dna50/franklingosling.pdf.
- ↑ "Original X-ray diffraction image". Osulibrary.oregonstate.edu. 30 Ocak 2009 tarihinde kaynağından arşivlendi. http://web.archive.org/web/20090130111849/http://osulibrary.oregonstate.edu:80/specialcollections/coll/pauling/dna/pictures/franklin-typeBphoto.html. Erişim tarihi: 2011-02-06.
- ↑ Wilkins M.H.F., A.R. Stokes A.R. & Wilson, H.R. (1953). "Molecular Structure of Deoxypentose Nucleic Acids" (PDF). Nature 171 (4356): 738–740. DOI:10.1038/171738a0. PMID 13054693. http://www.nature.com/nature/dna50/wilkins.pdf.
- ↑ The Nobel Prize in Physiology or Medicine 1962 Nobelprize .org Accessed 22 December 06
- ↑ Brenda Maddox (23 January 2003). "The double helix and the 'wronged heroine'" (PDF). Nature 421 (6921): 407–408. DOI:10.1038/nature01399. PMID 12540909. http://www.biomath.nyu.edu/index/course/hw_articles/nature4.pdf.
- ↑ Crick, F.H.C. On degenerate templates and the adaptor hypothesis (PDF). genome.wellcome.ac.uk (Lecture, 1955). Accessed 22 Dec 2006
- ↑ Meselson M, Stahl F (1958). "The replication of DNA in Escherichia coli". Proc Natl Acad Sci USA 44 (7): 671–82. DOI:10.1073/pnas.44.7.671. PMID 16590258.
- ↑ The Nobel Prize in Physiology or Medicine 1968 Nobelprize.org Accessed 22 Dec 06
Bibliyografya
- Clayton, Julie. (Ed.). 50 Years of DNA, Palgrave MacMillan Press, 2003. ISBN 978-1-4039-1479-8
- Judson, Horace Freeland. The Eighth Day of Creation: Makers of the Revolution in Biology, Cold Spring Harbor Laboratory Press, 1996. ISBN 978-0-87969-478-4 (İngilizce)
- Olby, Robert. The Path to The Double Helix: Discovery of DNA, first published in October 1974 by MacMillan, with foreword by Francis Crick; ISBN 978-0-486-68117-7(İngilizce)
- Matt Ridley. Francis Crick: Discoverer of the Genetic Code (Eminent Lives) HarperCollins Publishers; 192 pp, ISBN 978-0-06-082333-7 2006 (İngilizce)
- Rose, Steven. The Chemistry of Life, Penguin, ISBN 978-0-14-027273-4. (İngilizce)
- Watson, James D. and Francis H.C. Crick. A structure for Deoxyribose Nucleic Acid (PDF). Nature 171, 737–738, (1953). (İngilizce)
- Watson, James D. DNA: The Secret of Life ISBN 978-0-375-41546-3. (İngilizce)
- Watson, James D. The Double Helix: A Personal Account of the Discovery of the Structure of DNA (Norton Critical Editions). ISBN 978-0-393-95075-5 (İngilizce)
- Watson, James D. "Avoid boring people and other lessons from a life in science" (2007) New York: Random House. ISBN 978-0-375-41284-4 (İngilizce)
- Calladine, Chris R.; Drew, Horace R.; Luisi, Ben F. and Travers, Andrew A. Understanding DNA, Elsevier Academic Press, 2003. ISBN 978-0-12-155089-9 (İngilizce)
Dış bağlantılar
- Tübitak-DNA
- http://www.dna.tubitak.gov.tr/
- Genomlarla eğlence: Mikoçiğner DNA Bulmacası
- DNA testi (belgesel videolar)
- DNA verileri ve Millî DNA veri bankası kanun tasarısı ; Türkiye Tabibler Derneği ve Adli Bilimciler Derneği'nin bunun hakkındaki görüşleri.
- DNA testinin İspat hukuku Açısından Değerlendirilmesi
- Olay Yerinden DNA analizi için Biyolojik Örnek Toplama
- Double helix: 50 years of DNA, Nature Nature dergisinin DNA'nın yapısının keşfinin 50 yıldönümü özel sayısı. (İngilizce)
- Elektron microskobu altında DNA (İngilizce)
- DNA'nın bükülerek kromozomları oluşturması (animasyon)
- İki animasyon: 1) DNA'dan kromozomların oluşması ve 2) DNA ikileşmesi.
- DISPLAR: DNA binding site prediction on protein (İngilizce)
- DNA klonlaması hakkında temel animasyonlu rehber (İngilizce)
- DNA the Double Helix Game: Resmî Nobel Ödülü Web sitesinden DNA ikileşmesiyle ilgili oyun. (İngilizce)
- DNA Lab, kolayca temin edilebilecek araç ve gereçlerle buğdaydan nasıl DNA elde edilebileceğini gösteriyor. (İngilizce video)

